Einführung
die Replikation im Zellkern von eukaryotischen Zellen beschäftigt, drei DNA-Polymerasen: – polymerase α, δ und ε (Hubscher et al., 2002; Pavlov et al., 2006b; Kunkel und Burger, 2008; Loeb und Monnat, 2008; Burger, 2009; Pavlov und Shcherbakova, 2010; Lange et al., 2011)., Die DNA-Synthese ist direktional und verläuft von 5′ bis 3′, wobei ein nukleophiler Angriff auf das α-Phosphat eines Nukleotids durch die 3 ‚ OH eines Primers zum Einbau eines Nukleosidmonophosphats und zur Freisetzung von Pyrophosphat führt (Steitz, 1999). Alle DNA-Polymerasen benötigen einen Primer und ein freies 3 ‚ OH, um die DNA-Synthese durchzuführen, und pol α ist keine Ausnahme. Pol α ist ein Heterotetramer, der aus zwei Primase-Untereinheiten und zwei Polymerase-Untereinheiten besteht., Die Primase-Untereinheiten initiieren die DNA-Replikation durch Synthese kurzer (7-12 Ribonukleotide) RNA-Primer, die dann durch Polymerase α erweitert werden (Pellegrini, 2012). DNA-Polymerase δ und ε verlängern die von pol α erzeugten Primer genau und prozessiv (Kunkel, 2004, 2011; Pellegrini, 2012). In Hefe hat sich gezeigt, dass DNA-Polymerase δ für die DNA-Synthese des verzögerten Strangs essentiell ist, während pol ε hauptsächlich am führenden Strang zu funktionieren scheint (Pursell et al., 2007; Nick Mcelhinny et al., 2008; Kunkel, 2011; Georgescu et al., 2014)., Im Gegensatz dazu liegt in den Mitochondrien die Replikation in der Verantwortung einer einzigen Polymerase, der DNA-Polymerase γ (Lee et al., 2009).
DNA-Polymerasen sind unterteilt in sieben Familien (A, B, C, D, X, Y und RT). In Eukaryoten gehören die drei nuklear replikativen DNA-Polymerasen zufällig zur B-Familie (Burgers et al., 2001; Patel und Loeb, 2001). Es gibt nun Kristalle Strukturen aller drei replikativen DNA-Polymerasen aus Hefe, die erstmals einen Vergleich ihrer gemeinsamen Strukturelemente sowie eine Untersuchung ihrer einzigartigen Eigenschaften ermöglichen (Swan et al., 2009; Perera et al.,, 2013; Hogg et al., 2014; Jain et al., 2014a). Alle drei replikativen DNA-Polymerasen sind multi-subunit Enzyme (Tabelle 1) (Johansson und Macneill, 2010; Pavlov und Shcherbakova, 2010; Makarova et al., 2014). Der Schwerpunkt dieser Überprüfung liegt auf ihrer katalytischen Domäne oder Untereinheit A.
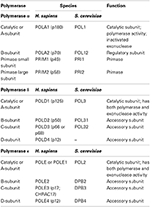
Tabelle 1. Eukaryotische DNA-Polymerasen sind Multi-Untereinheiten-Enzyme.,
Gesamtstruktur der B-Familienpolymerasen
Alle DNA-Polymerasen teilen eine gemeinsame Polymerasefalte, die mit einer menschlichen rechten Hand verglichen wurde, die aus drei Subdomänen besteht: Finger, Handfläche und Daumen (Steitz, 1999; Patel und Loeb, 2001). Die Palme, eine hochkonservierte Falte aus vier antiparallelen β-Strängen und zwei Helices, beherbergt zwei streng konservierte katalytische Aspartate in Motiv A, DXXLYPS und Motiv C, DTDS (Delarue et al., 1990; Braithwaite und Ito, 1993)., Diese RRM-ähnliche Falte wird von einer sehr großen Gruppe von Enzymen geteilt, einschließlich DNA – und RNA-Polymerasen, Reverse-Transkriptasen, CRISPR-Polymerase und sogar Reverse (3′-5′) Transferasen wie Thg1 (Anantharaman et al., 2010; Hyde et al., 2010). Im Gegensatz dazu weisen die Subdomänen Daumen und Finger eine wesentlich größere strukturelle Vielfalt auf (Steitz, 1999). Die Finger erfahren eine Konformationsänderung bei der Bindung von DNA und dem korrekt ankommenden Nukleotid. Durch diese Bewegung können Rückstände in der Finger-Subdomäne mit dem Nukleotid im entstehenden Basenpaar in Kontakt kommen., Der Daumen hält den DNA-Duplex während der Replikation und spielt eine Rolle in der Prozessivität (Doublié und Ellenberger, 1998; Doublié et al., 1999).
Eukaryotische DNA-Polymerasen α, δ und ε teilen sich die Homologie mit vielen archaealen, bakteriellen, bakteriophagen und viralen Polymerasen (Delarue et al., 1990; Braithwaite und Ito, 1993; Franklin et al., 2001; Firbank et al., 2008; Wang und Yang, 2009)., Koonin und seine Mitarbeiter trugen eine detaillierte phylogenetische Analyse von archaealen DNA-Polymerasen und ihrer Beziehung zu eukaryotischen Polymerasen in dieser Ausgabe von Frontiers in Microbiology zu Polymerasen Makarova et al. (2014).
Alle Polymerasen der B-Familie bestehen aus fünf Subdomänen: den Fingern, dem Daumen und der Handfläche (oben beschrieben), die den Kern des Enzyms bilden, sowie einer Exonukleasedomäne und einer N-terminalen Domäne (NTD) (Franklin et al., 2001; Xia und Königsberg, 2014) (Abbildung 1; Tabelle S1)., Die Exonukleasedomäne trägt eine 3′-5′ Korrekturleseaktivität, die falsch korporative Nukleotide entfernt. Die aktive Exonuklease-Stelle befindet sich 40-45 Å von der aktiven Polymerase-Stelle entfernt. Die NTD scheint keine katalytische Aktivität zu haben. In pol δ umfasst die NTD drei Motive: Eines hat eine Topologie, die einer OB-Falte ähnelt, ein einzelsträngiges DNA-Bindungsmotiv und ein anderes trägt ein RNA-Bindungsmotiv (RNA Recognition Motif oder RRM) (Swan et al., 2009). In Bakteriophagen T4 verringern Mutationen in der NTD die Expression der Polymerase (Hughes et al., 1987)., In RB69 und T4 bindet die gp43-Polymerase ihre eigene Messenger-RNA, vermutlich durch die NTD und unterdrückt Translation (Petrov et al., 2002), was bei pol δ nicht der Fall zu sein scheint (Swan et al., 2009). Neue Daten zeigen, dass die NTD durch ihre Wechselwirkungen mit anderen Domänen eine Rolle bei der Stabilität und Treue der Polymerase spielt (Li et al., 2010; Prindle et al., 2013) (siehe unten).
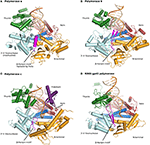
Abbildung 1. Ternäre Komplexe der Polymerasen α, δ, ε und RB69 gp43 werden aus identischen Orientierungen zum Vergleich dargestellt., Daumen (grün) und Finger (dunkelblau) greifen die Duplex-Nukleinsäure (Primer in Beige, Schablone in Orange) gegen die Palmdomäne (rot). Die N-Terminal-Domäne erscheint in Gold, angrenzend an die 3′ -5 ‚ Exonuklease-Domäne (Cyan). (A) Polymerase α (PDBID 4FYD) bindet einen RNA/DNA-Hybrid, wobei die breite, flache Nebenrille der A-Form-DNA nahe dem Daumen sichtbar ist. Die 3‘ -5 ‚ Exonuklease-Domäne ist frei von Aktivität. Eine helikale Region (Magenta) in der inaktivierten Exonukleasedomäne stabilisiert das 5 ‚ Ende der Schablone., (B) Polymerase δ (PDBID 3IAY) beherbergt ein großes β-Haarnadelmotiv (magenta), das beim Umschalten des Primerstrangs von der aktiven Polymerase-Stelle zur aktiven Exonuklease-Stelle im Falle eines Korrekturlesens wichtig ist. (C) Polymerase ε (PDBID 4M8O) besitzt eine einzigartige P-Domäne (Purpur), die der Polymerase eine erhöhte Prozessivität verleiht. Interessanterweise ist das β-Haarnadelmotiv in pol ε verkümmert., (D) Die Erhaltung der DNA-Polymerasefalte der Familie B und der Domänenorganisation ist offensichtlich, wenn das Modellenzym aus Bakteriophagen RB69 gp43 (PDBID 2OZS) zusammen mit den drei eukaryotischen replikativen Polymerasen betrachtet wird. Die Domänenabgrenzung für jede Polymerase ist in Tabelle S1 angegeben. Die Abbildung wurde mit PyMOL (The PyMOL Molecular Graphics System, Version 1.5.0.4 Schrödinger, LLC.).
Es ist bekannt, dass alle DNA-Polymerasen der Säugetier-B-Familie zwei cysteinreiche Metallbindungsstellen (CysA und CysB) in ihrer C-terminalen Domäne (CTD) aufweisen (Abbildung 2)., CysA wird als Zinkbindungsstelle vermutet, während CysB ein Eisenschwefelcluster ist (Netz et al., 2012). Der Verlust des Clusters in der CTD von Hefepol δ wirkt sich negativ auf Wechselwirkungen mit seiner Zubehör-B-Untereinheit aus (Sanchez Garcia et al., 2004). Es wurde gezeigt, dass das Zinkbindungsmotiv für die Wechselwirkung von pol δ mit seinem Prozessivitätsfaktor PCNA wichtig ist (Netz et al., 2012).

Abbildung 2. Schematische Darstellung der drei Saccharomyces cerevisiae replikative DNA-Polymerasen α, δ und ε., Zum Vergleich wird die DNA-Polymerase aus Bakteriophagen RB69 gezeigt.
DNA-Polymerase α
Die katalytische Untereinheit der DNA-polymerase-α besteht aus 1468 Aminosäuren (Tabelle 2). Das für die Kristallisation konzipierte Proteinkonstrukt wurde am N – und C-Termini abgeschnitten (Reste 349-1258) und es fehlt daher die CTD und ihr Cluster (Abbildung 2). Das Konstrukt wurde unversehrt kristallisiert, in einem binären Komplex mit einem DNA/RNA-Hybrid-Oligonukleotid und in einem ternären Komplex mit DNA/RNA und ankommendem Nukleotid (Perera et al., 2013) (Abbildung 1).,

Tabelle 2. Zusammenstellung von DNA-Polymerasen der B-Familie bekannter Struktur.
Ein Mechanismus zum Lösen der Polymerase
Das in den Kristallen eingefangene RNA / DNA-Oligonukleotid nimmt erwartungsgemäß eine A-Form-Konformation an. Die Daumendomäne interagiert sowohl über hydrophobe Kontakte als auch über polare Wechselwirkungen mit dem RNA-Primer mehrfach (Perera et al., 2013)., Versuche in Lösung haben gezeigt, dass die Ausdehnung des RNA-Primers durch pol α auf 10-12 Nukleotide begrenzt ist, was einer Umdrehung einer Helix entspricht. Diese Beobachtung veranlasste die Autoren, einen Mechanismus für die Beendigung der Primersynthese durch pol α vorzuschlagen, bei dem der Verlust spezifischer Wechselwirkungen zwischen dem Daumen und der RNA dazu führen würde, dass sich die Polymerase von dem DNA/RNA-Oligonukleotid löst und eine Übergabe an eine replikative Polymerase ermöglicht.,
Bewegungen im Palmbereich können die Translokation von Pol α erleichtern
Nachdem das Enzym in drei Zuständen (apo, binär und ternär) kristallisiert war, konnten die Autoren alle drei Strukturmodelle überlagern. Pol α ist die einzige DNA-Polymerase der eukaryotischen Familie B, für die alle drei Zustände in einer Kristallstruktur eingefangen wurden. Die strukturelle Überlagerung ergab, dass zusätzlich zu den gut dokumentierten Bewegungen der Finger – und Daumen-Subdomänen, die die Substratbindung und den Nukleotidyltransfer begleiten, die Palm-Subdomäne selbst einer strukturellen Umlagerung unterliegt (Perera et al.,, 2013). Die Autoren schlagen vor, dass die verschiedenen Konformationen der Palmdomäne die Translokation von pol α entlang und über den RNA/DNA-Duplex hinaus erleichtern könnten. Wie oben erwähnt, wird der Verlust von Kontakten zum RNA-Strang vorhergesagt, um die Freisetzung von Primer auszulösen, der dann zur Verlängerung durch pol δ oder ε verfügbar wird.,
Eine andere Proteinfalte in der inaktivierten Exonuklease-Subdomäne
Die Korrekturleseaktivität wird in pol α aufgrund von Mutationen in allen vier Carboxylaten (Asp114/Glu116/Asp222/Asp327 in RB69 gp43 entsprechen Ser542/Gln544/Tyr644 / Asn757 in einer strukturbasierten Ausrichtung) abgeschafft (Tabelle 2). Darüber hinaus wird das β-Haarnadelmotiv, das in den meisten Polymerasen der B-Familie gefunden wird (Rückstände 246-267 in RB69 gp43), durch einen helikalen Bereich in pol α ersetzt (Rückstände 667-676; 681-693) (Hogg et al., 2007)., Die β-Haarnadel ist Teil der Exonukleasedomäne und wurde in T4-und RB69-Pols gezeigt, dass sie an der Partitionierung des DNA-Primers zwischen der Polymerase und der aktiven Exonuklease-Stelle beteiligt ist (Reha-Krantz, 1988; Stocki et al., 1995; Hogg et al., 2007). In Ermangelung von Korrekturlesen ist es nicht verwunderlich, dass dieses Motiv nicht in polaris beibehalten wurde. Rückstände His 684 und Phe 685 der helikalen Region in pol α stapeln sich mit einer Thymin-und Guaninbasis an den Positionen -3 und -2 im ungepaarten 5 ‚ end der Schablone (Perera et al., 2013)., Somit übernimmt in pol α der dem β-Haarnadelmotiv entsprechende Bereich eine andere Falte (Helices vs. β-Stränge) und eine andere Funktion (Stabilisierung des ungepaarten Bereichs des Schablonenstrangs, anstatt eine aktive Standortumschaltung zu erleichtern). Da pol α keine Korrekturleseaktivität aufweist, stellt sich die Frage, ob die kurzen Oligonukleotide korrigiert werden und wenn ja, durch welche DNA-Polymerase. Es scheint, dass das Korrekturlesen der von pol α synthetisierten Primer von pol δ durchgeführt wird (Pavlov et al., 2006a).,
DNA-Polymerase δ
Die humane Polymerase δ besteht aus vier Untereinheiten, während Saccharomyces cerevisiae drei hat (Gerik et al., 1998; Liu et al., 2000) (Tabelle 1). Zusätzlich zu seiner Funktion bei der DNA-Replikation spielt δ nachweislich eine Rolle bei der DNA-Reparatur und-Rekombination (Hubscher et al., 2002; Lee et al., 2012; Tahirov, 2012). P12, die kleinste Untereinheit in der menschlichen Polarität und auch die Untereinheit, die in knospender Hefe nicht zu sehen ist, wird als Reaktion auf DNA-Schäden abgebaut (Lee et al., 2014). Die katalytische Untereinheit der Hefe pol δ (POL3) besteht aus 1097 Rückständen., Das für die Kristallisation verwendete Konstrukt umfasst die Rückstände 67-985 und fehlt somit der CTD (Abbildung 1; Tabelle 2).
Ein drittes Metallion in der aktiven Polymerase-Stelle
Die Palmdomäne enthält drei konservierte Carboxylate (Asp608, Asp762 und Asp764). Die beiden katalytischen Aspartate Asp608 und Asp764 berühren zwei Metallionen (Ca2+) an der aktiven Polymerase-Stelle, die durch 3,7 Å getrennt sind. Interessanterweise wurde ein drittes Metall beobachtet, das durch das γ-Phosphat des ankommenden Nukleotids und Glu802 koordiniert wurde, wobei Glu800 in der Nähe war., Die Mutierung beider Glutamate zu Alanin ergab eine Polymerase-Variante mit reduzierter Einbaueffizienz für korrekte und falsche Nukleotide (Swan et al., 2009). An diesen Aminosäurepositionen weisen pol α und pol ε ebenfalls Carboxylatreste auf (pol δ Glu800/Glu802 entsprechen pol α Asp1033/Asp1035 und pol ε Glu945/Asp947). Ob diese Carboxylate bei pol α und ε eine ähnliche Rolle spielen, muss noch untersucht werden.,
High Fidelity and Proofreading
Human pol δ ist eine High-Fidelity-Polymerase, die die Nukleotidyltransferreaktion mit einer Fehlerfrequenz von 1 pro 22.000 katalysiert (Schmitt et al., 2009). Korrekturlesen steigert die Wiedergabetreue der Polymerase um den Faktor 10-100 (Mcculloch und Kunkel, 2008; Prindle et al., 2013). Pol δ beherbergt eine aktive Polymerase-und Exonuklease-Stelle, die durch etwa 45 Å getrennt ist (Swan et al., 2009). DNA-Polymerasen mit Korrekturleseaktivität sind in der Lage, falsch korporative Nukleotide zu erfassen, indem sie die kleine Rille von Basenpaaren jenseits der Insertionsstelle kontaktieren., Das Protein interagiert mit universellen Wasserstoffbindungsakzeptoren an der N3-und O2-Position von Purinen bzw., 1976; Doublié et al., 1998; Franklin et al., 2001). Diese Wasserstoffbindungskontakte bleiben erhalten, wenn das Basispaar eine Watson-Crick-Geometrie annimmt und im Falle einer Nichtübereinstimmung verloren geht. In RB69 gp43 erstrecken sich die Kontakte zu den ersten beiden Basispaaren über das entstehende Basispaar hinaus (Franklin et al., 2001; Hogg et al., 2004, 2005). Die Kontakte sind in pol δ viel umfangreicher und erstrecken sich nach dem Einsetzen auf fünf Basispaare (Swan et al.,, 2009), was zu seiner hohen Treue beitragen könnte.
Wie oben erwähnt, spielt das β-Haarnadelsegment aus der Exonukleasedomäne eine entscheidende Rolle bei der Aufteilung der DNA zwischen Polymerisations – und Korrekturlesestellen in T4 – und RB69-Pols (Stocki et al., 1995; Hogg et al., 2007). In RB69 gp43 nimmt das β-Haarnadelmotiv unterschiedliche Konformationen an, abhängig davon, ob der Komplex mit unbeschädigter DNA erhalten wurde (Franklin et al., 2001; Zahn et al., 2007) oder DNA, die einen Schaden enthält (Freisinger et al., 2004; Hogg et al., 2004)., Es wurde vollständig visualisiert, indem sowohl die Primer-als auch die Schablonenstränge in einem Komplex mit Thyminglykol kontaktiert wurden (Aller et al., 2011). In ähnlicher Weise ragt die β-Haarnadel in pol δ in die Hauptnut der DNA und wirkt als Keil zwischen doppelsträngiger DNA und dem einzelsträngigen 5 ‚ end des Schablonenstrangs, der durch zwei aromatische Rückstände Phe441 und Tyr446 stabilisiert wird (Abbildung 1) (Swan et al., 2009). Die Position der β-Haarnadel stimmt mit einer Rolle bei der aktiven Standortumschaltung überein.,
Interdomainkontakte und Treue
Mutationen, die an Krebs beteiligt sind, finden sich hauptsächlich im Exonukleasebereich von pol δ und ε, was die kritische Rolle des Korrekturlesens bei der Senkung der Inzidenz von Mutationen unterstreicht (Church et al., 2013; Henninger und Pursell, 2014). Eine Mutation in menschlichen kolorektalen Krebszellen lokalisiert sich auf die Fingerdomäne, R689W.Die analoge Mutation in Hefe (R696W) führt zu einem Mutatorphänotyp (Daee et al., 2010)., Eine Mutation in der Nähe von Arg696 im hochkonservierten Motiv B der Finger-Subdomäne von Hefepol δ (A699Q) führt ebenfalls zu einem Mutatorphänotyp. Dieser Bereich der Finger befindet sich in unmittelbarer Nähe der NTD. Durch die Mutierung von Met540 des NTD zu Alanin wird der Mutatorphänotyp von A699Q abgeschafft, was zeigt, dass Wechselwirkungen zwischen den Fingern und dem NTD die Wiedergabetreue der Polymerase beeinflussen können (Prindle et al., 2013)., Ähnlich in T4-und RB69-Pols ist das NPL-Kernmotiv, das Rückstände aus den N-terminalen und Palm-Domänen beinhaltet, in Kontakt mit der Fingerdomäne und wurde gezeigt, dass es Polymerase-DNA-Komplexe stabilisiert (Li et al., 2010).
DNA-Polymerase ε
Die katalytische Untereinheit der DNA-Polymerase ε ist das Produkt eines sehr großen Gens(2222 Aminosäuren in Hefe; 2286 beim Menschen) und nur an dritter Stelle nach Polymerase π (ebenfalls ein Mitglied der B-Familie) und pol θ, einer Familie Eine Polymerase (3130 bzw.,, 2011; Hogg und Johansson, 2012) (Abbildung 1; Tabelle 2). Pol ε ist doppelt so groß wie pol δ und besteht aus zwei Tandem-Polymerase/Exonuklease-Regionen. Das N-Terminal-Segment beherbergt sowohl Polymerase-als auch Korrekturlesen, während das C-Terminal-Segment inaktiviert ist. Die beiden Exonuklease-Polymerase-Module sind distanziert verwandt (Tahirov et al., 2009). Obwohl angenommen wird, dass das inaktivierte Segment während der Replikation eine strukturelle Rolle spielt, konnten zwei Gruppen katalytisch aktive pol ε-Konstrukte kristallisieren (Rückstände 1-1228; 1-1187), denen das gesamte C-Terminalmodul fehlte (Hogg et al.,, 2014; Jain et al., 2014a). Beide Kristallstrukturen waren von einem ternären Komplex der Polymerase, DNA-Primer / Schablone und des ankommenden Nukleotids.
Eine neuartige Prozessivitätsdomäne, die von der Palmdomäne ausgeht
Pol ε unterscheidet sich von pol δ dadurch, dass es für eine hohe Prozessivität keine DNA-Gleitklemme PCNA benötigt (Hogg und Johansson, 2012). Die Palmdomäne von pol ε ist wesentlich größer (380 Rückstände) als die von pol α oder δ (175 bzw., Die jüngsten pol ε-Kristallstrukturen zeigten, dass Insertionen in der Palmdomäne kollektiv eine neue Domäne bilden, die aus drei β-Strängen und zwei Helices besteht (Rest 533-555; 682-760) (Hogg et al., 2014; Jain et al., 2014a) (Abbildung 1; Tabelle S1). Das Löschen von Rückständen 690-751 führte zu einer Variante mit verminderter Polymeraseaktivität. Darüber hinaus beeinflussten mutierende positiv geladene Rückstände (His748, Arg749 und Lys751), die sich in der Nähe des Phosphatrückgrats befanden, die Prozessivität des Enzyms (Hogg et al., 2014)., Die zusätzliche Domäne, die von der Palme stammt, wurde daher nach ihrer Funktion als Prozessivitäts-oder P-Domäne bezeichnet. Die Basis der P-Domäne beherbergt eine Metallbindungsstelle (siehe unten) (Hogg et al., 2014; Jain et al., 2014a,b).
Ein Eisenschwefelcluster innerhalb der Polymerasedomäne
Unerwartet zeigten Lösungsstudien, dass die katalytische Untereinheit der Hefepolymerase ε selbst einen Cluster innerhalb ihrer Polymerasefalte enthält (Jain et al., 2014b), zusätzlich zu dem Cluster in der CTD (Abbildung 2; Tabelle 2)., Der zweite Cluster innerhalb von pol ε legt nahe, dass diese Polymerase empfindlicher auf oxidativen Stress reagieren kann (Jain et al., 2014b). Die Kristallstrukturen von pol ε zeigten jedoch keinen Cluster im Polymerase-Bereich (Hogg et al., 2014; Jain et al., 2014a; Zahn-und Doublié, 2014). Zwei der Cysteinrückstände sind in den Strukturmodellen ungeordnet und die resultierende Metallbindungsstelle scheint Zink zu binden (Hogg et al., 2014; Jain et al., 2014a). Die Substitution von a durch ein nicht natives Zink in metallbindenden Proteinen ist nicht ungewöhnlich (Netz et al., 2012) da Cluster labil sind., Die Visualisierung des Polymerasebereichs von pol ε kann anaerobe Bedingungen erfordern.
Ein kurzes β-Haarnadelmotiv in der Exonukleasedomäne
In jeder DNA-Polymerase, die sowohl Polymerase-als auch Exonukleaseaktivitäten enthält, befindet sich die gebundene DNA im Gleichgewicht zwischen den beiden aktiven Zentren (Beechem et al., 1998). Die Konzentration des ankommenden Nukleotids und das Vorhandensein einer beschädigten Base oder eines Fehlpaares sind zwei Faktoren, die den Transfer von DNA von der Polymerase-Aktivierungsstelle zur Korrekturlesen-aktiven Stelle beeinflussen., Polymerasen überwachen die kleinere Nutseite der neu gebildeten Basispaare und interagieren mit den universellen H-Bondakzeptoren O3 und N2, um auf Nichtübereinstimmungen zu prüfen (Seeman et al., 1976; Franklin et al., 2001). Ein einzigartiges Merkmal von pol ε ist der Kontakt zur Hauptnutenseite des entstehenden Basispaares über einen Rückstand aus der Exonukleasedomäne Tyr431. Eine weitere Analyse ist gerechtfertigt, um die potenzielle Rolle dieses Tyrosins bei der hohen Wiedergabetreue von pol ε aufzuklären.,
In pol δ fügt sich das β-Haarnadelsegment in die DNA ein und fungiert als Keil zwischen einzelsträngiger und doppelsträngiger DNA (Swan et al., 2009). In E. coli DNA pol II verschiebt die Insertion eines β-Fasses die Position der β-Haarnadel so, dass die Polymerisation gegenüber dem Korrekturlesen begünstigt wird (Wang und Yang, 2009). Diese Modifikation ermöglicht vermutlich, dass diese Polymerase eine Translesionsyntheseerweiterung durchführt., Da pol ε eine genaue DNA-Polymerase ist, würde die Annahme vor der Kenntnis der Kristallstruktur sein, dass die β-Haarnadel näher an der von pol δ sein sollte als die von E. coli Pol II. Überraschenderweise ist das β-Haarnadel-Motiv in pol ε abgeschnitten, zu kurz, um die DNA zu kontaktieren (Abbildung 1). Welches Proteinmotiv könnte also den aktiven Standortwechsel bei der Erkennung eines Fehlpaares erleichtern? Die P-Domäne ist aufgrund ihrer Kontakte zu Primer-und Vorlagensträngen ein guter Kandidat; Rückstände aus der P-Domäne könnten Replikationsfehler erkennen und somit dazu beitragen, den aktiven Standortwechsel zu erleichtern.,
Schlussfolgerungen
Alle drei eukaryotischen replikativen DNA-Polymerasen verwenden eine gemeinsame B-Familienfalte, und jede Polymerase hat modifizierte Strukturelemente integriert, die einzigartig und auf die spezifische Funktion jeder Polymerase zugeschnitten sind (z. B. die Zugabe der Prozessivitätsdomäne in pol ε, einer prozessiven Polymerase, die keine PCNA verwendet, oder der modifizierten Region, die das 5 ‚ Ende der Vorlage in pol α berührt, einer Polymerase ohne Korrekturleseaktivität). Die Falte der B-Familie Polymerasen ist gut geeignet für High-Fidelity, replikative Polymerasen., Überraschenderweise wird es aber auch von Translesion-Polymerasen verwendet. Eukaryotische Polycine (oder REV3L) ist eine 353-kDa-Polymerase, die bei der Translesionsynthese wirkt und die Tumorigenese zu unterdrücken scheint (Wittschieben et al., 2010; Lange et al., 2011; Zahn et al., 2011; Hogg und Johansson, 2012; Sharma et al., 2013). Die Struktur von E. coli Pol II zeigte Modifikationen in der NTD, die die Position der β-Haarnadel der Exonukleasedomäne und damit die Aufteilung der DNA zwischen den Polymerisations-und Korrekturlesestellen beeinflussen (Wang und Yang, 2009)., Die Struktur von polycin kann ähnliche Anpassungen aufweisen, die die Faltung von High-Fidelity-replikativen Polymerasen verändern, um das Enzym weniger treu und in der Lage zu machen, Translesionsynthese durchzuführen.
Interessenkonflikterklärung
Die Autoren erklären, dass die Untersuchung ohne kommerzielle oder finanzielle Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Anerkennung
Diese Arbeit wurde durch einen Zuschuss der National Institutes of Health (NCI R01 CA 52040) unterstützt.
















Schreibe einen Kommentar