wprowadzenie
replikacja w jądrze komórek eukariotycznych wykorzystuje trzy polimerazy DNA: polimeraza α, δ i ε (Hubscher et al., 2002; Pavlov et al., 2006b; Kunkel and Burgers, 2008; Loeb and Monnat, 2008; Burgers, 2009; Pavlov and Shcherbakova, 2010; Lange et al., 2011)., Synteza DNA jest kierunkowa i przebiega od 5′ do 3′, gdzie nukleofilowy atak na fosforan α nukleotydu przez 3 ' OH podkładu powoduje włączenie monofosforanu nukleozydów i uwolnienie pirofosforanu (Steitz, 1999). Wszystkie polimerazy DNA wymagają podkładu i wolnego 3 ' OH do przeprowadzenia syntezy DNA, a pol α nie jest wyjątkiem. POL α jest heterotetramerem złożonym z dwóch podjednostek primazy i dwóch podjednostek polimerazy., Podjednostki primazy inicjują replikację DNA poprzez syntezę krótkich (7-12 rybonukleotydów) primerów RNA, które następnie są rozszerzane przez polimerazę α (Pellegrini, 2012). Polimeraza DNA δ i ε wydłużają primery generowane przez pol α w sposób dokładny i procesywny (Kunkel, 2004, 2011; Pellegrini, 2012). W drożdżach wykazano, że polimeraza DNA δ jest niezbędna do syntezy DNA opóźnionej nici, podczas gdy POL ε wydaje się działać głównie na wiodącej nici (Pursell et al., 2007; Nick Mcelhinny et al., 2008; Kunkel, 2011; Georgescu et al., 2014)., W przeciwieństwie do replikacji mitochondriów jest odpowiedzialny za jedną polimerazę sole, polimeraza DNA γ (Lee et al., 2009).
polimerazy DNA są zgrupowane w siedmiu rodzinach (A, B, C, D, X, Y i RT). U eukariotów trzy nuklearne replikacyjne polimerazy DNA należą do rodziny B (Burgers et al., 2001; Patel i Loeb, 2001). Obecnie istnieją struktury kryształów wszystkich trzech replikacyjnych polimeraz DNA z drożdży, które po raz pierwszy pozwalają na porównanie ich wspólnych elementów strukturalnych ,a także badanie ich unikalnych cech (Swan et al., 2009; Perera et al.,, 2013; Hogg et al., 2014; Jain et al., 2014a). Wszystkie trzy replikacyjne polimerazy DNA są enzymami podjednostkowymi (Tabela 1) (Johansson and Macneill, 2010; Pavlov and Shcherbakova, 2010; Makarova et al., 2014). Głównym celem tej recenzji jest ich katalityczna domena lub podjednostka A.
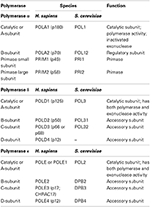
Tabela 1. Eukariotyczne polimerazy DNA są enzymami wielozadaniowymi.,
ogólna struktura polimeraz rodziny B
wszystkie polimerazy DNA mają wspólny fałd polimerazy, który porównano do ludzkiej prawej ręki, składający się z trzech subdomen: palców, dłoni i kciuka (Steitz, 1999; Patel i Loeb, 2001). Palma, wysoce konserwowany fałd składający się z czterech antyparallel β nici i dwie helisy, zawiera dwa ściśle konserwowane katalityczne aspartany znajdujące się w motif A, DXXLYPS i motif C, DTDS (Delarue et al., 1990; Braithwaite i Ito, 1993)., Ten fałd podobny do RRM jest dzielony przez bardzo dużą grupę enzymów, w tym polimerazy DNA i RNA, odwrotne transkryptazy, polimerazę CRISPR, a nawet odwrotne (3′-5′) transferazy, takie jak Thg1 (Anantharaman et al., 2010; Hyde i in., 2010). Natomiast subdomeny kciuka i palców wykazują znacznie większą różnorodność strukturalną (Steitz, 1999). Palce ulegają zmianie konformacyjnej po wiązaniu DNA i prawidłowym przychodzącym nukleotydzie. Ruch ten pozwala pozostałościom w subdomenie palców na kontakt z nukleotydem w rodzącej się parze zasad., Kciuk utrzymuje dupleks DNA podczas replikacji i odgrywa rolę w procesie (Doublié and Ellenberger, 1998; Doublié et al., 1999).
eukariotyczne polimerazy DNA α, δ i ε mają wspólną homologię z wieloma archaealnymi, bakteryjnymi, bakteriofagami i wirusowymi polimerazami (Delarue et al., 1990; Braithwaite and Ito, 1993; Franklin et al., 2001; Firbank et al., 2008; Wang i Yang, 2009)., Koonin i współpracownicy przyczynili się do szczegółowej analizy filogenetycznej archaealnych polimeraz DNA i ich związku z polimerazami eukariotycznymi w tym zagadnieniu granic w mikrobiologii poświęconej polimerazom Makarova et al. (2014).
wszystkie polimerazy rodziny B składają się z pięciu subdomen: palców, kciuka i dłoni (opisanych powyżej) stanowiących rdzeń enzymu, a także domeny egzonukleazy i domeny N-terminalnej (NTD) (Franklin et al., 2001; Xia i Królewiec, 2014) (Rysunek 1; Tabela S1)., Domena egzonukleazy prowadzi aktywność korekcyjną 3′-5′, która usuwa błędne nukleotydy. Miejsce aktywne egzonukleazy znajduje się w odległości 40-45 Å od miejsca aktywnego polimerazy. NTD wydaje się być pozbawione aktywności katalitycznej. W pol δ NTD składa się z trzech motywów: jeden ma topologię przypominającą fałd OB, jednoniciowy motyw wiązania DNA, a drugi nosi motyw wiązania RNA (RNA Recognition Motif lub RRM) (Swan et al., 2009). W bakteriofagach T4, mutacje w NTD zmniejszają ekspresję polimerazy (Hughes et al., 1987)., W RB69 i T4, gp43 polimeraza wiąże swój własny posłaniec RNA, przypuszczalnie poprzez NTD i represje translacji (Petrov et al., 2002), co wydaje się nie mieć miejsca w przypadku Polki (Swan et al., 2009). Nowe dane wskazują, że NTD odgrywa rolę w stabilności i wierności polimerazy poprzez jej interakcje z innymi domenami (Li et al., 2010; Prindle et al., 2013) (zob. poniżej).
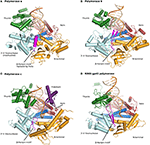
Rysunek 1. Trójskładnikowe kompleksy polimeraz α, δ, ε i rb69 gp43 są zilustrowane z identycznych orientacji dla porównania., Domeny kciuka (zielone) i palców (ciemnoniebieskie) chwytają dupleksowy kwas nukleinowy (podkład pokazany w kolorze beżowym, szablon w kolorze pomarańczowym) względem domeny dłoni (czerwony). Domena N-terminalna występuje w złocie, sąsiaduje z domeną egzonukleazy 3′ -5 ' (cyjan). (A) polimeraza α (PDBID 4FYD) wiąże hybrydę RNA / DNA, w której szeroki, płytki niewielki rowek DNA formy a jest widoczny w pobliżu kciuka. Domena egzonukleazy 3′-5′ jest pozbawiona aktywności. Region spiralny (magenta) w inaktywowanej domenie egzonukleazy stabilizuje 5 ' End szablonu., B) polimeraza δ (PDBID 3) zawiera duży motyw spinki β (magenta), który jest ważny w przełączaniu nici gruntowej z miejsca aktywnego polimerazy na miejsce aktywne egzonukleazy w przypadku korekty. (C) polimeraza ε (PDBID 4M8O) posiada unikalną domenę P (fioletową), która nadaje polimerazie zwiększoną procesowość. Co ciekawe, motyw spinki β zanika w polszczyźnie., (D) zachowanie rodziny B składanie polimerazy DNA, i organizacji domeny, jest oczywiste, gdy model enzymu z bakteriofaga rb69 gp43 (PDBID 2ozs)jest postrzegany wraz z trzema eukariotic replikacyjnych polimeraz. Oznaczenie domeny dla każdej polimerazy podano w tabeli S1. Rysunek został wykonany za pomocą PyMOL (The pymol Molecular Graphics System, Version 1.5.0.4.).
wszystkie polimerazy DNA rodziny B są znane z dwóch miejsc wiązania metali bogatych w cysteinę (Cisa i CysB) w ich domenie terminalnej C (CTD) (ryc. 2)., Uważa się, że CISA jest miejscem wiązania cynku, podczas gdy CysB jest klastrem siarki żelaza (Netz et al., 2012). Utrata klastra w CTD drożdży POL δ negatywnie wpływa na interakcje z jego dodatkową podjednostką B (Sanchez Garcia et al., 2004). Wykazano, że motyw wiązania cynku jest ważny dla interakcji pol δ z jego czynnikiem procesywności, PCNA (Netz et al., 2012).

Rysunek 2. Schemat trzech replikacyjnych polimeraz DNA Saccharomyces cerevisiae α, δ i ε., Dla porównania przedstawiono polimerazę DNA z bakteriofagów RB69.
polimeraza DNA α
podjednostka katalityczna polimerazy DNA α składa się z 1468 aminokwasów (Tabela 2). Konstrukcja białkowa przeznaczona do krystalizacji została obcięta na N-I C-termini (pozostałości 349-1258) i dlatego brakuje CTD i jego klastra (ryc. 2). Konstrukcja została skrystalizowana bez połączenia, w dwuskładnikowym kompleksie z hybrydowym OLIGONUKLEOTYDEM DNA / RNA oraz w trójskładnikowym kompleksie z DNA / RNA i nadchodzącym nukleotydem (Perera et al., 2013) (Rys. 1).,

Tabela 2. Kompilacja polimeraz DNA z rodziny B o znanej strukturze.
Mechanizm odłączania polimerazy
oligonukleotyd RNA / DNA wychwycony w kryształach przyjmuje konformację w postaci A, zgodnie z oczekiwaniami. Domena kciuka angażuje się w wiele interakcji z podkładem RNA, zarówno poprzez kontakty hydrofobowe, jak i interakcje polarne (Perera et al., 2013)., Eksperymenty w roztworze wykazały, że rozszerzenie RNA przez pol α jest ograniczone do 10-12 nukleotydów, co stanowi jeden obrót helisy. Ta obserwacja doprowadziła autorów do zaproponowania mechanizmu zakończenia syntezy podkładu przez Pol α, w którym utrata specyficznych interakcji między kciukiem a RNA spowodowałaby odłączenie polimerazy od OLIGONUKLEOTYDU DNA/RNA i umożliwienie ręki replikacyjnej polimerazie.,
ruchy w domenie Palmowej mogą ułatwić translokację Pol α
po krystalizacji enzymu w trzech stanach (apo, binarnym i trójkowym) pozwoliły autorom na nałożenie wszystkich trzech modeli strukturalnych. Pol α jest jedyną eukariotyczną polimerazą DNA rodziny B, dla której wszystkie trzy stany zostały uchwycone w strukturze krystalicznej. Superpozycja strukturalna ujawniła, że oprócz dobrze udokumentowanych ruchów subdomen palców i kciuka towarzyszących wiązaniu substratów i transferowi nukleotydów, subdomena dłoni sama ulega strukturalnej przegrupowaniu (Perera et al.,, 2013). Autorzy proponują, że różne konformacje domeny Palmowej mogłyby ułatwić translokację POL α wzdłuż i poza dupleksem RNA/DNA. Jak wspomniano powyżej, przewiduje się, że utrata styków do nici RNA spowoduje uwolnienie podkładu, który następnie staje się dostępny do rozszerzenia przez pol δ lub ε.,
inny fałd białkowy w inaktywowanej subdomenie egzonukleazy
aktywność korekcji zostaje zniesiona w pol α, z powodu mutacji we wszystkich czterech karboksylanach (Asp114/Glu116/Asp222/Asp327 w rb69 gp43 odpowiada ser542/Gln544/Tyr644/Asn757 w wyrównaniu opartym na strukturze) (Tabela 2). Ponadto Motyw β-spinki do włosów znaleziony w większości polimeraz z rodziny B (pozostałości 246-267 w rb69 gp43) zastępuje się regionem spiralnym w pol α (pozostałości 667-676; 681-693) (Hogg et al., 2007)., Spinka β jest częścią domeny egzonukleazy i wykazano, że w T4 i rb69 pols bierze udział w podziale DNA primer między polimerazę i miejsce aktywne egzonukleazy (Reha-Krantz, 1988; Stocki et al., 1995; Hogg et al., 2007). Wobec braku korekty nie dziwi fakt, że motyw ten nie został zachowany w języku polskim. Pozostałości His 684 i Phe 685 regionu spiralnego w stosie POL α z zasadą tyminą i guaniną, odpowiednio, w pozycjach -3 i -2 W niesparowanym 5 ' Endzie szablonu (Perera et al., 2013)., Tak więc w pol α region odpowiadający motywowi β-spinki przyjmuje inną fałdę (helisy vs. nici β) i inną funkcję (stabilizując niesparowany region nici szablonu, zamiast ułatwiać aktywną zmianę miejsca). Ponieważ pol α jest pozbawiony aktywności korekcyjnej, powstaje pytanie, czy krótkie oligonukleotydy są korygowane, a jeśli tak, to za pomocą której polimerazy DNA. Wydaje się, że korekta podkładów syntetyzowanych przez pol α jest wykonywana przez Pol δ (Pavlov et al., 2006a).,
polimeraza DNA δ
ludzki pol δ składa się z czterech podjednostek, podczas gdy Saccharomyces cerevisiae ma trzy (Gerik et al., 1998; Liu et al., 2000) (Tabela 1). Oprócz funkcji w replikacji DNA wykazano, że pol δ odgrywa rolę w naprawie i rekombinacji DNA (Hubscher et al., 2002; Lee et al., 2012; Tahirov, 2012). P12, najmniejsza podjednostka w ludzkiej pol δ, a także podjednostka, która nie jest widoczna w drożdżach budujących, ulega degradacji w odpowiedzi na uszkodzenie DNA (Lee et al., 2014). Podjednostka katalityczna drożdży POL δ (POL3) składa się z 1097 reszt., Konstrukcja stosowana do krystalizacji zawiera pozostałości 67-985, a zatem brakuje CTD (Rysunek 1; Tabela 2).
trzeci jon metalu w miejscu aktywnym polimerazy
domena Palmowa zawiera trzy zachowane karboksylaty (Asp608, Asp762 i Asp764). Dwa katalityczne aspartany, Asp608 i Asp764, kontaktują się z dwoma jonami metali (Ca2+) w miejscu aktywnym polimerazy oddzielonym od siebie 3,7 Å. Z kolei w pobliżu glu800 zaobserwowano trzeci metal koordynowany przez γ fosforanu przychodzącego nukleotydu i Glu802., Mutowanie obu glutaminianów do alaniny dało wariant polimerazy o zmniejszonej skuteczności inkorporacji zarówno dla prawidłowych, jak i nieprawidłowych nukleotydów (Swan et al., 2009). W tych pozycjach aminokwasowych POL α i POL ε mają również pozostałości karboksylowe (pol δ glu800/Glu802 odpowiadają POL α Asp1033/Asp1035, a POL ε glu945/Asp947). Czy te karboksylany odgrywają podobną rolę w pol α i ε pozostaje do zbadania.,
High Fidelity and Proofreading
Human POL δ jest polimerazą o wysokiej wierności, katalizującą reakcję przeniesienia nukleotydów z częstotliwością błędu 1 na 22 000 (Schmitt et al., 2009). Korekta zwiększa wierność polimerazy o współczynnik 10-100 (Mcculloch and Kunkel, 2008; Prindle et al., 2013). Pol δ Zawiera miejsce aktywne polimerazy i egzonukleazy, oddzielone o około 45 Å (Swan et al., 2009). Polimerazy DNA o działaniu korekcyjnym są w stanie wyczuć błędne nukleotydy poprzez kontakt z mniejszym rowkiem par zasad poza miejscem wstawiania., Białko współdziała z uniwersalnymi akceptorami wiązań wodorowych w pozycjach N3 i O2 puryn i pirymidyn, odpowiednio (Seeman et al., 1976; Doublié et al., 1998; Franklin et al., 2001). Te kontakty wiązania wodorowego są zachowane, gdy para zasad przyjmuje geometrię Watsona-Cricka i traci w przypadku niedopasowania. W rb69 gp43, kontakty rozciągają się do dwóch pierwszych par bazowych poza rodzącą się parą bazową (Franklin et al., 2001; Hogg et al., 2004, 2005). Styki są znacznie bardziej rozbudowane w polaryzacji, rozszerzając się do pięciu par bazalnych po wstawieniu (Swan et al.,, 2009), co może przyczynić się do jego wysokiej wierności.
jak wspomniano powyżej, segment β-spinka z domeny egzonukleazy odgrywa kluczową rolę w podziale DNA między polimeryzacji i korekty miejsc w T4 i rb69 pols (Stocki et al., 1995; Hogg et al., 2007). W rb69 gp43 Motyw β-Spinka przyjmuje różne konformacje, w zależności od tego, czy kompleks został uzyskany z nieuszkodzonego DNA (Franklin et al., 2001; Zahn et al., 2007) lub DNA zawierającego uszkodzenie (Freisinger et al., 2004; Hogg et al., 2004)., Został w pełni zwizualizowany kontaktując się zarówno z pasmami podkładu, jak i szablonu w kompleksie z glikolem tyminowym (Aller et al., 2011). Podobnie, β Spinka w pol δ wystaje w głównym rowku DNA i działa jako klin między dwuniciowym DNA i jednoniciowym 5 ' End nici szablonu, który jest stabilizowany przez dwie pozostałości aromatyczne Phe441 i Tyr446 (Rysunek 1) (Swan et al., 2009). Położenie spinki β jest zgodne z rolą w aktywnej zmianie miejsca.,
Interdomain Kontakty i wierność
mutacje zaangażowane w raka są głównie w domenie egzonukleazy POL δ i ε, podkreślając krytyczną rolę korekty w obniżaniu częstości występowania mutacji (Church et al., 2013; Henninger and Pursell, 2014). Jedna mutacja w ludzkich komórkach raka jelita grubego lokalizuje się do domeny palców, R689W. analogiczna mutacja w drożdży (r696w) powoduje fenotyp mutatora (Daee et al., 2010)., Mutacja w pobliżu Arg696 w silnie zachowanym motywie B subdomeny palców drożdży POL δ (A699Q) również skutkuje fenotypem mutatora. Ten region palców znajduje się w pobliżu NTD. Mutacja Met540 z NTD do alaniny znosi fenotyp mutatora A699Q, ilustrując, że interakcje między palcami i NTD mogą wpływać na wierność polimerazy (Prindle et al., 2013)., Podobnie w T4 i rb69 Pols motyw rdzenia NPL, który obejmuje pozostałości z domen N-końcowych i palm, jest w kontakcie z domeną palców i wykazano, że stabilizuje kompleksy polimerazy-DNA (Li et al., 2010).
polimeraza DNA ε
podjednostka katalityczna polimerazy DNA ε jest produktem bardzo dużego genu (2222 aminokwasy w drożdży; 2286 u ludzi) i jest tylko trzecia pod względem wielkości po polimerazie ζ (również członek rodziny B) i POL θ, polimerazy rodziny a (3130 i 2590 aminokwasów, odpowiednio u ludzi) (Lange et al.,, 2011; Hogg and Johansson, 2012) (rysunek 1; Tabela 2). POL ε jest dwa razy większy od POL δ i składa się z dwóch regionów polimerazy tandemowej/egzonukleazy. Segment N-końcowy zawiera zarówno polimerazę, jak i korektę, podczas gdy segment C-końcowy jest inaktywowany. Dwa moduły egzonukleazy-polimerazy są odlegle spokrewnione (Tahirov et al., 2009). Chociaż przypuszcza się, że inaktywowany segment odgrywa rolę strukturalną podczas replikacji, dwie grupy były w stanie krystalizować katalitycznie aktywne konstrukcje POL ε (pozostałości 1-1228; 1-1187) pozbawione całego modułu terminala C (Hogg et al.,, 2014; Jain et al., 2014a). Obie struktury krystaliczne składały się z trójskładnikowego kompleksu polimerazy, podkładu/szablonu DNA i przychodzącego nukleotydu.
nowa domena Procesywności pochodząca z domeny Palmowej
Pol ε różni się od POL δ tym, że nie wymaga przesuwnego zacisku DNA PCNA dla wysokiej procesywności (Hogg and Johansson, 2012). Domena palm POL ε jest znacznie większa (380 reszt) niż domena POL α lub δ (odpowiednio 175 i 203 reszt)., Niedawne struktury krystaliczne pol ε ujawniły, że wstawki w domenie Palmowej wspólnie tworzą nową domenę składającą się z trzech nici β i dwóch Helis (pozostałości 533-555; 682-760) (Hogg et al., 2014; Jain et al., 2014a) (Rysunek 1; Tabela S1). Usunięcie pozostałości 690-751 spowodowało odmianę o zmniejszonej aktywności polimerazy. Ponadto mutacja dodatnio naładowanych pozostałości (His748, Arg749 i Lys751) znajdujących się w pobliżu szkieletu fosforanowego wpłynęła na processivity enzymu (Hogg et al., 2014)., Dodatkowa domena pochodząca z palmy została nazwana domeną processivity lub P, od jej funkcji. Podstawa domeny P zawiera miejsce wiązania metalu (patrz poniżej) (Hogg et al., 2014; Jain et al., 2014a, b).
Klaster siarki żelaza w domenie polimerazy
nieoczekiwanie badania roztworu wykazały, że podjednostka katalityczna polimerazy drożdży ε sama zawiera klaster w swoim foldzie polimerazy (Jain et al., 2014b), oprócz klastra w CTD (Rys. 2; Tabela 2)., Drugi klaster w obrębie pol ε sugeruje, że polimeraza ta może być bardziej wrażliwa na stres oksydacyjny (Jain et al., 2014b). Struktury krystaliczne Polinezji nie ujawniły jednak gromady w domenie polimerazy (Hogg et al., 2014; Jain et al., 2014a; Zahn i Doublié, 2014). Dwie pozostałości cystein są nieuporządkowane w modelach strukturalnych, a powstałe miejsce wiązania metalu wydaje się wiązać cynk (Hogg et al., 2014; Jain et al., 2014a). Zastąpienie a przez nienaturalnego cynku w białkach wiążących metale nie jest niczym niezwykłym (Netz et al., 2012) jako clusters are labile., Wizualizacja wewnątrz domeny polimerazy pol ε może wymagać warunków beztlenowych.
Krótki motyw spinki do włosów β w domenie egzonukleazy
W dowolnej polimerazie DNA, która zawiera zarówno aktywność polimerazy, jak i egzonukleazy, związany DNA znajduje się w równowadze między dwoma aktywnymi ośrodkami (Beechem et al., 1998). Stężenie przychodzącego nukleotydu oraz obecność uszkodzonej Zasady lub mispairu to dwa czynniki wpływające na przeniesienie DNA z miejsca aktywacji polimerazy do miejsca aktywnego., Polimerazy monitorują niewielką stronę rowka nowo utworzonych par zasadowych i współdziałają z uniwersalnymi akceptorami wiązania H, O3 i N2, jako sposób sprawdzania niedopasowania (Seeman et al., 1976; Franklin et al., 2001). Unikalną cechą pol ε jest kontakt z główną stroną rowka rodzącej się pary zasad przez pozostałość z domeny egzonukleazy, Tyr431. Dalsza analiza jest uzasadniona, aby wyjaśnić potencjalną rolę tej tyrozyny w wysokiej wierności pol ε.,
w pol δ segment β-Spinka wstawia się w DNA i działa jako klin między jednoniciowym i dwuniciowym DNA (Swan et al., 2009). W E. coli DNA pol II, wstawianie beczki β przesuwa położenie spinki β w taki sposób, że polimeryzacja jest preferowana nad korektą (Wang and Yang, 2009). Ta modyfikacja prawdopodobnie pozwala tej polimerazie na przeprowadzenie rozszerzenia syntezy translesion., Ponieważ POL ε jest dokładną polimerazą DNA, założenie przed poznaniem struktury krystalicznej byłoby takie, że Spinka β powinna być bliżej spinki δ niż E. coli POL II. co zaskakujące, motyw spinki β w Pol ε jest ścięty, zbyt krótki, aby zetknąć się z DNA (ryc. 1). Który motyw białka może ułatwić aktywną zmianę miejsca po wykryciu błędu? Domena P jest dobrym kandydatem, ze względu na kontakty zarówno z pasmami primer, jak i template; pozostałości z domeny P mogą wykrywać błędy replikacji, a tym samym ułatwiać aktywną zmianę strony.,
wnioski
wszystkie trzy eukariotyczne replikacyjne polimerazy DNA wykorzystują wspólną fałdę rodziny B, a każda polimeraza zawiera zmodyfikowane elementy strukturalne, które są unikalne i dostosowane do specyficznej funkcji każdej polimerazy (na przykład dodanie domeny procesywności w Pol ε, polimerazie procesywnej, która nie używa PCNA, lub zmodyfikowany region kontaktujący się z 5 ' End szablonu w Pol α, polimerazie pozbawionej aktywności korekty). Fałd polimeraz rodziny B jest dobrze przystosowany do polimeraz replikacyjnych o wysokiej wierności., Ale zaskakująco, jest również używany przez polimerazy translesion. Eukariotyczna polymeraza (lub REV3L) jest polimerazą o mocy 353 kDa, która działa w syntezie translacji i wydaje się hamować tumorigenezę (Wittschieben et al., 2010; Lange et al., 2011; Zahn et al., 2011; Hogg and Johansson, 2012; Sharma et al., 2013). Struktura E. coli Pol II ujawniła zmiany w NTD, które wpływają na położenie spinki β domeny egzonukleazy, a tym samym na podział DNA między miejsca polimeryzacji i korekty (Wang and Yang, 2009)., Struktura polimerazy polimerowej może wykazywać podobne zmiany, które zmieniają fałd stosowany przez polimerazy replikacyjne o wysokiej wierności, aby enzym był mniej wierny i zdolny do wykonywania syntezy translesionalnej.
Oświadczenie o konflikcie interesów
autorzy oświadczają, że badanie zostało przeprowadzone w przypadku braku jakichkolwiek relacji handlowych lub finansowych, które mogłyby być interpretowane jako potencjalny konflikt interesów.
praca ta była wspierana przez grant z National Institutes of Health (NCI R01 CA 52040).
















Dodaj komentarz