zakończenie projektu ludzkiego genomu w 2003 zapoczątkował nową erę szybkiej, niedrogiej i dokładnej analizy genomu—zwanej next Generation Sequencing (NGS). NGS opiera się na technologiach” sekwencjonowania pierwszej generacji”, aby uzyskać dokładne i opłacalne wyniki sekwencjonowania.
Fred Sanger zsekwencjonował pierwszy cały genom DNA, wirus Fage ?X174, w 1977 roku., W tym samym roku Sanger opracował przyszłą podstawę ery genomu: technologię sekwencjonowania DNA. Jego technika wykorzystywała metodę zakończenia łańcucha. Jest to obecnie postrzegane jako „technologia pierwszej generacji” sekwencjonowania genomu.
ponieważ sekwencjonowanie Sangera (lub metoda zakończenia łańcucha) jest pierwszą generacją technologii sekwencjonowania, zrozumienie tego jest bardzo ważne. Projekt Human Genome rozpoczął się od technologii sekwencjonowania Sangera.,
metoda sekwencjonowania Sangera
metoda sekwencjonowania Sangera polega na dideoksynukleotydach (ddNTPs),rodzaju trójfosforanów deoksynukleozydów (dNTPs), które nie posiadają grupy hydroksylowej 3′ i mają atom wodoru zamiast . Gdy te zasady wiążą się z rosnącą sekwencją DNA, przerywają replikację, ponieważ nie mogą wiązać innych zasad. Aby przeprowadzić sekwencjonowanie Sangera, dodajesz podkłady do roztworu zawierającego informacje genetyczne, które mają zostać zsekwencjonowane, a następnie dzielisz roztwór na cztery reakcje PCR., Każda reakcja zawiera mieszankę a z dNTP z jednym z czterech nukleotydów podstawionych ddNTP (grupy A, T, G I C ddntp). Na końcu PCR, każda z czterech reakcji daje produkty PCR o różnych długościach, ponieważ replikacja jest losowo zakończona. Uruchamiając próbki na żelu z 4 torami, możesz złożyć sekwencję, ponieważ każda sekwencja została odtworzona z tego samego oryginalnego materiału. Oto przykład, w którym ddNTPs są pogrubione, a dNTPs Nie:
twoja Sekwencja to ATGCTCAG.,
Twoje cztery reakcje dają:
reakcja z ddATP: A, ATGCTCA, ATGCTCAG
reakcja z DDGTP: ATG, ATGCTCAG
reakcja z DDCTP: ATGC, ATGCTC, ATGCTCAG
reakcja z DDTTP: AT, ATGCT, ATGCTCAG
wszystkie reakcje po uruchomieniu żelu wyglądałyby mniej więcej tak Olwen Reina):
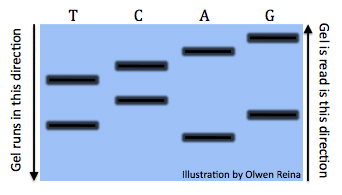
każdy zespół oznacza kod o różnej długości. Na przykład, zespół jest prawo pod „A” symbolizuje sekwencję: „ATGCTCA”
nadal zdezorientowany?
wyobraźmy sobie grę imprezową. Gra jest zgadywanką., Oto jak to jest grane:
myślisz o numerze i grupa musi go odgadnąć. Najtrudniejsze jest to, że liczba ma długość 200 cyfr. Czytasz cyfry numeru w głowie, nie wydając dźwięku. Co jakiś czas ktoś ci przerywa, a ty mówisz mu o jednej cyfrze, którą właśnie myślałeś i gdzie ona jest w sekwencji 200. Za każdym razem, gdy ktoś ci przerywa, musisz zacząć od nowa. Wyjeżdżasz po kilku godzinach, a grupa musi ustalić 200-cyfrowy numer., Muszą poskładać informacje, które im podałeś, na przykład 25. liczba to 5, 40. Liczba to 0 i tak dalej. Korzystając z informacji z przerw, mogą powtórzyć numer, który Ci Dali.
choć brzmi to jak lamest gry na świecie, to działa bardzo dobrze dla sekwencjonowania!
Niestety jest powolny, drogi i (wcześniej) opiera się na materiałach radioaktywnych. To popchnęło naukowców do opracowania nowych i lepszych form sekwencjonowania genomu.,
technologie sekwencjonowania następnej generacji
największe postępy w sekwencjonowania genomu zostały zwiększenie szybkości i dokładności, co powoduje zmniejszenie siły roboczej i kosztów. Szybkość jest dzięki równoległej analizie i technologii wysokiej przepustowości. Jest to różnica między tym, że 4-latek czyta Moby Dicka, a tym, że każdy z nich daje paragraf Bandzie kierunków dramatycznych i prosi je o przeczytanie na raz.
Maszyny do sekwencjonowania znacznie się poprawiły, odkąd Sanger rozwinął swoją metodę., W 1987 roku Applied Biosystems jako pierwszy wprowadził automatyczną maszynę sekwencjonującą o nazwie AB370. Maszyna opierała się na metodzie zwanej elektroforezą kapilarną, która wykorzystywała metodę zakończenia łańcucha Sangera bez konieczności użycia żelu.
Za pomocą metody sekwencjonowania Sangera, produkty PCR o różnych długościach są tworzone, a następnie oddzielane w zależności od ich wielkości. Rozmiar jest mierzony przez całkowity ujemny ładunek produktu PCR. Im bardziej ujemny ładunek, tym dłuższy fragment., Weźmy powyższy przykład włączenia ddNTP i wyobraźmy sobie, że pogrubione litery to ddNTPs z dołączonym fluorochromem. Gdy fragmenty są przyciągane w kierunku dodatniej elektrody kapilary( patrz rysunek poniżej), przechodzą przez wiązkę lasera, która wyzwala błysk światła z fluorochromu dołączonego do ddntp, który jest charakterystyczny dla typu podstawowego (na przykład zielony dla A, Żółty Dla T, niebieski dla G, czerwony dla C). W ten sposób Genom jest uważnie czytany.
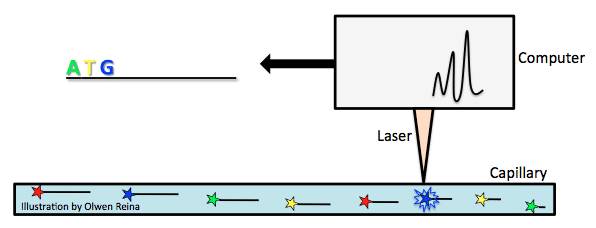
największą różnicą była tutaj szybkość i koszt., AB370 mógł wykryć 96 baz w jednym czasie, 500 000 baz dziennie i miał długość odczytu 600 baz przy użyciu analizy równoległej i konfiguracji wysokiej przepustowości. Ta forma sekwencjonowania stał się głównym narzędziem do zakończenia projektu ludzkiego genomu.
jak NGS porównuje się do sekwencjonowania pierwszej generacji?
NGS charakteryzuje się lepszą dokładnością i szybkością, ale także zmniejszoną siłą roboczą i kosztami. Nigdy nie było czasu, w którym sekwencja genomu była tak tania, wygodna lub prosta., Prawdopodobnie największą poprawą było opracowanie analizy równoległej, która zwiększyła szybkość sekwencjonowania.
jedyne co nas teraz spowalnia to interpretacja wyników!

czy to ci pomogło? Następnie podziel się z siecią.
















Dodaj komentarz