Introduzione
La replicazione nel nucleo delle cellule eucariotiche impiega tre DNA polimerasi: polimerasi α, δ e ε (Hubscher et al., 2002; Pavlov et al., 2006b; Kunkel e hamburger, 2008; Loeb e Monnat, 2008; Hamburger, 2009; Pavlov e Shcherbakova, 2010; Lange et al., 2011)., La sintesi del DNA è direzionale e procede da 5 ‘a 3’, dove l’attacco nucleofilo all’α fosfato di un nucleotide da parte del 3’OH di un primer provoca l’incorporazione di un monofosfato nucleosidico e il rilascio di pirofosfato (Steitz, 1999). Tutte le DNA polimerasi richiedono un primer e un 3’OH libero per condurre la sintesi del DNA, e pol α non fa eccezione. Pol α è un eterotetramero composto da due subunità primasi e due subunità polimerasi., Le subunità della primasi iniziano la replicazione del DNA sintetizzando primer RNA brevi (7-12 ribonucleotidi), che vengono poi estesi dalla polimerasi α (Pellegrini, 2012). DNA polimerasi δ e ε allungano i primer generati da pol α in modo accurato e processivo (Kunkel, 2004, 2011; Pellegrini, 2012). Nel lievito, la DNA polimerasi δ ha dimostrato di essere essenziale per la sintesi del DNA del filamento in ritardo, mentre pol ε sembra funzionare principalmente nel filamento principale (Pursell et al., 2007; Nick Mcelhinny et al., 2008; Kunkel, 2011; Georgescu et al., 2014)., Al contrario, nella replicazione dei mitocondri è la responsabilità di una sola polimerasi, la DNA polimerasi γ (Lee et al., 2009).
Le DNA polimerasi sono raggruppate in sette famiglie (A, B, C, D, X, Y e RT). Negli eucarioti le tre DNA polimerasi replicative nucleari appartengono alla famiglia B (Burgers et al., 2001; Patel e Loeb, 2001). Ci sono ora strutture di cristalli di tutte e tre le DNA polimerasi replicative dal lievito, che consentono per la prima volta un confronto dei loro elementi strutturali condivisi e uno studio delle loro caratteristiche uniche (Swan et al., 2009; Perera et al.,, 2013; Hogg et al., 2014; Jain et al., 2014a). Tutte e tre le DNA polimerasi replicative sono enzimi multi-subunità (Tabella 1) (Johansson e Macneill, 2010; Pavlov e Shcherbakova, 2010; Makarova et al., 2014). L’obiettivo principale di questa recensione è sul loro dominio catalitico, o subunità A.
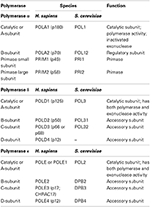
Tabella 1. Le DNA polimerasi eucariotiche sono enzimi multi-subunità.,
Struttura complessiva delle polimerasi della famiglia B
Tutte le DNA polimerasi condividono una piega comune della polimerasi, che è stata confrontata con una mano destra umana, composta da tre sottodomini: dita, palmo e pollice (Steitz, 1999; Patel e Loeb, 2001). Il palmo, una piega altamente conservata composta da quattro filamenti β antiparalleli e due eliche, ospita due aspartati catalitici strettamente conservati situati nel motivo A, DXXLYPS e nel motivo C, DTDS (Delarue et al., 1990; Braithwaite e Bra, 1993)., Questa piega simile a RRM è condivisa da un gruppo molto ampio di enzimi, tra cui DNA e RNA polimerasi, trascrittasi inverse, polimerasi CRISPR e persino transferasi inverse (3′-5′) come Thg1 (Anantharaman et al., 2010; Hyde et al., 2010). Al contrario, i sottodomini pollice e dita mostrano una diversità sostanzialmente più strutturale (Steitz, 1999). Le dita subiscono un cambiamento conformazionale su DNA legante e il nucleotide entrante corretto. Questo movimento consente ai residui nel sottodominio delle dita di entrare in contatto con il nucleotide nella coppia di basi nascente., Il pollice tiene il DNA duplex durante la replicazione e svolge un ruolo nella processività (Doublié ed Ellenberger, 1998; Doublié et al., 1999).
Le DNA polimerasi eucariotiche α, δ e ε condividono l’omologia con molte polimerasi archaeali, batteriche, batteriofaghe e virali (Delarue et al., 1990; Braithwaite e It, 1993; Franklin et al., 2001; Firbank et al., 2008; Wang e Yang, 2009)., Koonin e collaboratori hanno contribuito ad una dettagliata analisi filogenetica delle DNA polimerasi archaeal e del loro rapporto con le polimerasi eucariotiche in questo numero di Frontiers in Microbiology dedicato alle polimerasi Makarova et al. (2014).
Tutte le polimerasi della famiglia B sono composte da cinque sottodomini: le dita, il pollice e il palmo (descritti sopra) che costituiscono il nucleo dell’enzima, nonché un dominio esonucleasi e un dominio N-terminale (NTD) (Franklin et al., 2001; Xia e Konigsberg, 2014) (Figura 1; Tabella S1)., Il dominio esonucleasi svolge un’attività di correzione di bozze 3 ‘-5’, che rimuove i nucleotidi misincorporati. Il sito attivo dell’esonucleasi si trova a 40-45 Å dal sito attivo della polimerasi. L’NTD sembra essere privo di attività catalitica. In pol δ l’NTD comprende tre motivi: uno ha una topologia simile a una piega OB, un motivo di legame del DNA a filamento singolo, e un altro porta un motivo di legame dell’RNA (motivo di riconoscimento dell’RNA o RRM) (Swan et al., 2009). Nel batteriofago T4, le mutazioni nell’NTD diminuiscono l’espressione della polimerasi (Hughes et al., 1987)., In RB69 e T4, la polimerasi gp43 lega il proprio RNA messaggero, presumibilmente attraverso l’NTD e reprime la traduzione (Petrov et al., 2002), che non sembra essere il caso di pol δ (Swan et al., 2009). Nuovi dati indicano che l’NTD svolge un ruolo nella stabilità e nella fedeltà della polimerasi attraverso le sue interazioni con altri domini (Li et al., 2010; Prindle et al., 2013) (vedi sotto).
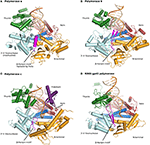
Figura 1. I complessi ternari delle polimerasi α, δ, ε e RB69 gp43 sono illustrati da orientamenti identici per il confronto., Il pollice (verde) e le dita (blu scuro) domini afferrare l’acido nucleico duplex (primer mostrato in beige, modello in arancione) contro il dominio di palma (rosso). Il dominio N-terminale appare in oro, adiacente al dominio 3′ -5 ‘ esonucleasi (ciano). (A) La polimerasi α (PDBID 4FYD) lega un ibrido RNA/DNA, dove il solco minore largo e poco profondo del DNA A-forma è evidente vicino al pollice. Il dominio esonucleasi 3 ‘ -5 ‘ è privo di attività. Una regione elicoidale (magenta) nel dominio inattivato dell’esonucleasi stabilizza il 5’end del modello., (B) La polimerasi δ (PDBID 3IAY) ospita un grande motivo a forcina β (magenta), che è importante nel passaggio del filo di primer dal sito attivo della polimerasi al sito attivo dell’esonucleasi in caso di correzione di bozze. (C) La polimerasi ε (PDBID 4M8O) esercita un dominio P unico (viola), che conferisce alla polimerasi una maggiore processività. È interessante notare che il motivo della forcella β è atrofizzato in pol ε., (D) La conservazione della piega della DNA polimerasi della famiglia B e l’organizzazione del dominio sono evidenti quando l’enzima modello del batteriofago RB69 gp43 (PDBID 2OZS) viene visualizzato insieme alle tre polimerasi replicative eucariotiche. La delineazione del dominio per ciascuna polimerasi è riportata nella Tabella S1. La figura è stata realizzata con PyMOL (il sistema grafico molecolare PyMOL, versione 1.5.0.4 Schrödinger, LLC.).
Tutte le DNA polimerasi della famiglia B dei mammiferi sono note per ospitare due siti di legame metallico ricco di cisteina (CysA e CysB) nel loro dominio C-terminale (CTD) (Figura 2)., Si presume che CysA sia un sito di legame allo zinco mentre CysB è un ammasso di zolfo di ferro (Netz et al., 2012). La perdita del cluster nel CTD del lievito pol δ influisce negativamente sulle interazioni con la sua subunità B accessoria (Sanchez Garcia et al., 2004). Il motivo di legame con lo zinco è stato dimostrato importante per l’interazione di pol δ con il suo fattore di processività, PCNA (Netz et al., 2012).

Figura 2. Diagramma schematico dei tre Saccharomyces cerevisiae replicative DNA polimerasi α, δ e ε., La DNA polimerasi del batteriofago RB69 viene mostrata per confronto.
DNA Polimerasi α
La subunità catalitica della DNA polimerasi α è composta da 1468 aminoacidi (Tabella 2). Il costrutto proteico progettato per la cristallizzazione è stato troncato ai termini N e C (residui 349-1258) e quindi manca del CTD e del suo cluster (Figura 2). Il costrutto è stato cristallizzato non legato, in un complesso binario con un oligonucleotide ibrido DNA/RNA e in un complesso ternario con DNA / RNA e nucleotide in entrata (Perera et al., 2013) (Figura 1).,

Tabella 2. Compilazione di DNA polimerasi della famiglia B di struttura nota.
Un meccanismo di disimpegno della polimerasi
L’oligonucleotide RNA / DNA catturato nei cristalli adotta una conformazione A-forma, come previsto. Il dominio del pollice si impegna in molteplici interazioni con il primer RNA, sia tramite contatti idrofobici che interazioni polari (Perera et al., 2013)., Esperimenti in soluzione hanno dimostrato che l’estensione del primer RNA da parte di pol α è limitata a 10-12 nucleotidi, che equivale a un giro di un’elica. Questa osservazione ha portato gli autori a suggerire un meccanismo per la terminazione della sintesi di primer da parte di pol α in cui la perdita di interazioni specifiche tra il pollice e l’RNA innescherebbe la polimerasi a disimpegnarsi dall’oligonucleotide DNA/RNA e consentire una mano a una polimerasi replicativa.,
I movimenti nel dominio del palmo possono facilitare la traslocazione di Pol α
Avendo cristallizzato l’enzima in tre stati (apo, binario e ternario) ha permesso agli autori di sovrapporre tutti e tre i modelli strutturali. Pol α è l’unica famiglia eucariotica B DNA polimerasi per la quale tutti e tre gli stati sono stati catturati in una struttura cristallina. La sovrapposizione strutturale ha rivelato che, oltre ai movimenti ben documentati delle dita e dei sottodomini del pollice che accompagnano il legame del substrato e il trasferimento nucleotidico, il sottodominio del palmo subisce un riarrangiamento strutturale (Perera et al.,, 2013). Gli autori propongono che le diverse conformazioni del dominio palm potrebbero facilitare la traslocazione di pol α lungo e oltre il duplex RNA/DNA. Come accennato in precedenza, si prevede che la perdita di contatti con il filamento di RNA inneschi il rilascio di primer, che diventa quindi disponibile per l’estensione di pol δ o ε.,
Una diversa piega proteica nel sottodominio dell’esonucleasi inattivata
L’attività di correzione delle bozze è abolita in pol α, a causa di mutazioni in tutti e quattro i carbossilati (Asp114/Glu116/Asp222/Asp327 in RB69 gp43 corrisponde a Ser542/Gln544/Tyr644 / Asn757 in un allineamento basato sulla struttura) (Tabella 2). Inoltre, il motivo β-tornante trovato nella maggior parte delle polimerasi della famiglia B (residui 246-267 in RB69 gp43) è sostituito da una regione elicoidale in pol α (residui 667-676; 681-693) (Hogg et al., 2007)., La forcella β fa parte del dominio dell’esonucleasi ed è stato dimostrato nei pol T4 e RB69 di partecipare al partizionamento del primer del DNA tra la polimerasi e il sito attivo dell’esonucleasi (Reha-Krantz, 1988; Stocki et al., 1995; Hogg et al., 2007). In assenza di attività di correzione di bozze non sorprende che questo motivo non sia stato mantenuto in pol α. Residui His 684 e Phe 685 della regione elicoidale in pol α stack con una base di timina e guanina, rispettivamente, alle posizioni -3 e -2 nel 5’end spaiato del modello (Perera et al., 2013)., Pertanto, in pol α la regione corrispondente al motivo β-tornante adotta una piega diversa (eliche rispetto a trefoli β) e una funzione diversa (stabilizzando la regione spaiata del filo del modello piuttosto che facilitare la commutazione attiva del sito). Poiché pol α è privo di attività di correzione di bozze, sorge la domanda se gli oligonucleotidi corti siano corretti e, in caso affermativo, con quale DNA polimerasi. Sembra che la correzione di bozze dei primer sintetizzati da pol α sia eseguita da pol δ (Pavlov et al., 2006a).,
La DNA Polimerasi δ
Human pol δ è composta da quattro subunità mentre Saccharomyces cerevisiae ne ha tre (Gerik et al., 1998; Liu et al., 2000) (Tabella 1). Oltre alla sua funzione nella replicazione del DNA pol δ ha dimostrato di svolgere un ruolo nella riparazione e nella ricombinazione del DNA (Hubscher et al., 2002; Lee et al., 2012; Tahirov, 2012). P12, la più piccola subunità nella pol δ umana e anche la subunità che non si vede nel lievito in erba, viene degradata in risposta al danno al DNA (Lee et al., 2014). La subunità catalitica del lievito pol δ (POL3) è composta da 1097 residui., Il costrutto utilizzato per la cristallizzazione comprende residui 67-985 e quindi manca del CTD (Figura 1; Tabella 2).
Un terzo Ion metallico nel sito attivo della polimerasi
Il dominio palm contiene tre carbossilati conservati (Asp608, Asp762 e Asp764). I due aspartati catalitici, Asp608 e Asp764, contattano due ioni metallici (Ca2+) nel sito attivo della polimerasi separati da 3,7 Å. Curiosamente è stato osservato un terzo metallo coordinato dal γ fosfato del nucleotide in arrivo e Glu802, con Glu800 nelle vicinanze., La mutazione di entrambi i glutammati in alanina ha prodotto una variante della polimerasi con ridotta efficienza di incorporazione sia per i nucleotidi corretti che per quelli errati (Swan et al., 2009). A queste posizioni aminoacidiche, pol α e pol ε hanno anche residui di carbossilato (pol δ Glu800/Glu802 corrispondono a pol α Asp1033/Asp1035 e pol ε Glu945/Asp947). Resta da studiare se questi carbossilati svolgono ruoli simili in pol α e ε.,
High Fidelity and Proofreading
Human pol δ è una polimerasi ad alta fedeltà, che catalizza la reazione di trasferimento nucleotidilico con una frequenza di errore di 1 per 22.000 (Schmitt et al., 2009). La correzione di bozze aumenta la fedeltà della polimerasi di un fattore di 10-100 (Mcculloch e Kunkel, 2008; Prindle et al., 2013). Pol δ ospita un sito attivo di polimerasi ed esonucleasi, separati da circa 45 Å (Swan et al., 2009). Le DNA polimerasi con attività di correzione di bozze sono in grado di rilevare nucleotidi misincorporati contattando la scanalatura minore delle coppie di basi oltre il sito di inserzione., La proteina interagisce con gli accettori universali del legame idrogeno nelle posizioni N3 e O2 delle purine e delle pirimidine, rispettivamente (Seeman et al., 1976; Doublié et al., 1998; Franklin et al., 2001). Questi contatti legame idrogeno sono conservati quando la coppia di base adotta una geometria Watson-Crick e perso in caso di mancata corrispondenza. In RB69 gp43, i contatti si estendono alle prime due coppie di basi oltre la coppia di basi nascente (Franklin et al., 2001; Hogg et al., 2004, 2005). I contatti sono molto più estesi in pol δ, estendendosi a cinque coppie di basi post-inserimento (Swan et al.,, 2009), che potrebbe contribuire alla sua alta fedeltà.
Come accennato in precedenza, il segmento β-tornante dal dominio esonucleasi svolge un ruolo critico nella partizione del DNA tra i siti di polimerizzazione e correzione di bozze nei pol T4 e RB69 (Stocki et al., 1995; Hogg et al., 2007). In RB69 gp43 il motivo β-tornante adotta diverse conformazioni, a seconda che il complesso sia stato ottenuto con DNA intatto (Franklin et al., 2001; Zahn et al., 2007) o DNA contenente un danno (Freisinger et al., 2004; Hogg et al., 2004)., È stato completamente visualizzato contattando sia il primer che i fili del modello in un complesso con glicole timinico (Aller et al., 2011). Allo stesso modo, la forcella β in pol δ sporge nella scanalatura principale del DNA e funge da cuneo tra il DNA a doppio filamento e il 5’end a singolo filamento del filamento del modello, che è stabilizzato da due residui aromatici Phe441 e Tyr446 (Figura 1) (Swan et al., 2009). La posizione della forcella β è coerente con un ruolo nella commutazione attiva del sito.,
Contatti Interdomain e Fidelity
Le mutazioni coinvolte nel cancro si trovano principalmente nel dominio dell’esonucleasi di pol δ e ε, sottolineando il ruolo critico della correzione di bozze nel ridurre l’incidenza delle mutazioni (Church et al., 2013; Henninger e Pursell, 2014). Una mutazione nelle cellule tumorali colorettali umane si localizza nel dominio delle dita, R689W. La mutazione analoga nel lievito (R696W) si traduce in un fenotipo mutatore (Daee et al., 2010)., Una mutazione in prossimità di Arg696 nel motivo altamente conservato B del sottodominio delle dita del lievito pol δ (A699Q) si traduce anche in un fenotipo mutatore. Questa regione delle dita si trova in prossimità dell’NTD. Mutare Met540 della NTD in alanina abolisce il fenotipo mutatore di A699Q, illustrando che le interazioni tra le dita e la NTD possono influenzare la fedeltà della polimerasi (Prindle et al., 2013)., Allo stesso modo in T4 e RB69 pols il motivo del nucleo NPL, che coinvolge residui dai domini N-terminale e palm, è in contatto con il dominio delle dita ed è stato dimostrato che stabilizza i complessi polimerasi-DNA (Li et al., 2010).
DNA Polimerasi ε
La subunità catalitica della DNA polimerasi ε è il prodotto di un gene molto grande (2222 amminoacidi nel lievito; 2286 nell’uomo), ed è solo la terza in termini di dimensioni dopo la polimerasi ζ (anch’essa membro della famiglia B) e pol θ, una famiglia A polimerasi (3130 e 2590 amminoacidi, rispettivamente, nell’uomo) (Lange et al.,, 2011; Hogg e Johansson, 2012) (Figura 1; Tabella 2). Pol ε è due volte più grande di pol δ ed è composto da due regioni tandem polimerasi/esonucleasi. Il segmento N-terminale ospita sia le attività di polimerasi che di correzione di bozze mentre il segmento C-terminale è inattivato. I due moduli esonucleasi-polimerasi sono lontanamente correlati (Tahirov et al., 2009). Sebbene si presume che il segmento inattivato svolga un ruolo strutturale durante la replicazione, due gruppi sono stati in grado di cristallizzare costrutti pol ε cataliticamente attivi (residui 1-1228; 1-1187) privi dell’intero modulo C-terminale (Hogg et al.,, 2014; Jain et al., 2014a). Entrambe le strutture cristalline erano di un complesso ternario della polimerasi, DNA primer / template e nucleotide in entrata.
Un nuovo dominio di processività che emana dal dominio Palm
Pol ε differisce da pol δ in quanto non richiede il DNA sliding clamp PCNA per un’alta processività (Hogg e Johansson, 2012). Il dominio di palma di pol ε è sostanzialmente più grande (380 residui) di quello di pol α o δ (175 e 203 residui, rispettivamente)., Le recenti strutture cristalline pol ε hanno rivelato che gli inserimenti nel dominio palm formano collettivamente un nuovo dominio costituito da tre filamenti β e due eliche (residui 533-555; 682-760) (Hogg et al., 2014; Jain et al., 2014a) (Figura 1; Tabella S1). L’eliminazione dei residui 690-751 ha prodotto una variante con ridotta attività della polimerasi. Inoltre, la mutazione dei residui caricati positivamente (His748, Arg749 e Lys751) situati nelle vicinanze della spina dorsale del fosfato ha influenzato la processività dell’enzima (Hogg et al., 2014)., Il dominio extra proveniente dal palmo è stato quindi chiamato processività o dominio P, dopo la sua funzione. La base del dominio P ospita un sito di legame metallico (vedi sotto) (Hogg et al., 2014; Jain et al., 2014a, b).
Un ammasso di ferro Zolfo all’interno del dominio della polimerasi
Inaspettatamente studi di soluzione hanno rivelato che la subunità catalitica della polimerasi ε del lievito contiene un ammasso all’interno della sua piega polimerasica (Jain et al., 2014b), oltre al cluster nel CTD (Figura 2; Tabella 2)., Il secondo cluster all’interno di pol ε suggerisce che questa polimerasi potrebbe essere più sensibile allo stress ossidativo (Jain et al., 2014b). Le strutture cristalline di pol ε, tuttavia, non hanno rivelato un cluster nel dominio della polimerasi (Hogg et al., 2014; Jain et al., 2014a; Zahn e Doublié, 2014). Due dei residui di cisteine sono disordinati nei modelli strutturali e il sito di legame metallico risultante sembra legare lo zinco (Hogg et al., 2014; Jain et al., 2014a). La sostituzione di a con uno zinco non nativo in proteine leganti il metallo non è insolita (Netz et al., 2012) come cluster sono labili., La visualizzazione all’interno del dominio della polimerasi di pol ε può richiedere condizioni anaerobiche.
Un breve motivo β-tornante nel dominio dell’esonucleasi
In qualsiasi DNA polimerasi che ospita sia le attività della polimerasi che dell’esonucleasi, il DNA legato è in equilibrio tra i due centri attivi (Beechem et al., 1998). La concentrazione di nucleotide in entrata e la presenza di una base danneggiata o mispair sono due fattori che influenzano il trasferimento del DNA dal sito di attivazione della polimerasi al sito attivo di correzione di bozze., Le polimerasi monitorano il lato minore della scanalatura delle coppie di basi appena formate e interagiscono con gli accettori universali del legame H, O3 e N2, come un modo per controllare i disallineamenti (Seeman et al., 1976; Franklin et al., 2001). Una caratteristica unica di pol ε è il contatto con il lato principale della scanalatura della coppia di basi nascente tramite un residuo del dominio dell’esonucleasi, Tyr431. Ulteriori analisi sono giustificate per chiarire il potenziale ruolo di questa tirosina nell’alta fedeltà di pol ε.,
In pol δ il segmento β-tornante si inserisce nel DNA e funge da cuneo tra DNA a filamento singolo e doppio filamento (Swan et al., 2009). In E. coli DNA pol II, l’inserimento di un barile β sposta la posizione della forcella β in modo tale che la polimerizzazione sia favorita rispetto alla correzione di bozze (Wang e Yang, 2009). Questa modifica presumibilmente consente a questa polimerasi di eseguire l’estensione della sintesi di traslazione., Poiché pol ε è una DNA polimerasi accurata, l’ipotesi prima della conoscenza della struttura cristallina sarebbe che la forcella β dovrebbe essere più vicina a quella di pol δ rispetto a quella di E. coli Pol II. Sorprendentemente, il motivo β-tornante in pol ε è troncato, troppo breve per contattare il DNA (Figura 1). Quale motivo proteico, quindi, potrebbe facilitare la commutazione del sito attivo al rilevamento di una mispair? Il dominio P è un buon candidato, a causa dei suoi contatti con entrambi i trefoli di primer e template; i residui del dominio P potrebbero rilevare errori di replica e quindi possono aiutare a facilitare la commutazione attiva del sito.,
Conclusioni
Tutti e tre eucariotica replicativa del DNA polimerasi uso comune B-famiglia piega, e ogni polimerasi ha incorporato modificato gli elementi strutturali che sono uniche e su misura per ogni polimerasi specifica funzione (per esempio, l’aggiunta di processivity dominio di pol ε, un processive polimerasi che non fa uso di PCNA, o modificato regione contattare il 5 end del modello in pol α, una polimerasi privo di attività di correzione. La piega delle polimerasi della famiglia B è adatta per polimerasi replicative ad alta fedeltà., Ma sorprendentemente, è anche usato dalle polimerasi di traslazione. La pol ζ eucariotica (o REV3L) è una polimerasi di 353 kDa che funziona nella sintesi della traslesione e sembra sopprimere la tumorigenesi (Wittschieben et al., 2010; Lange et al., 2011; Zahn et al., 2011; Hogg e Johansson, 2012; Sharma et al., 2013). La struttura di E. coli Pol II ha rivelato modifiche nell’NTD che influenzano la posizione della forcella β del dominio dell’esonucleasi, e quindi il partizionamento del DNA tra i siti di polimerizzazione e correzione di bozze (Wang e Yang, 2009)., La struttura di pol ζ può rivelare aggiustamenti simili, che alterano la piega impiegata dalle polimerasi replicative ad alta fedeltà per rendere l’enzima meno fedele e in grado di eseguire la sintesi della traslazione.
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Riconoscimento
Questo lavoro è stato sostenuto da una sovvenzione del National Institutes of Health (NCI R01 CA 52040).
















Lascia un commento