Il completamento del progetto Genoma umano nel 2003 ha inaugurato una nuova era di rapida, conveniente, e accurata analisi del genoma-chiamato Next Generation Sequencing (NGS). NGS si basa su tecnologie di “sequenziamento di prima generazione” per ottenere risultati di sequenziamento accurati e convenienti.
Fred Sanger sequenziato il primo genoma del DNA intero, il virus fago ?X174, nel 1977., Nello stesso anno, Sanger ha sviluppato la futura spina dorsale dell’era del genoma: la tecnologia di sequenziamento del DNA. La sua tecnica utilizzava il metodo di terminazione della catena. Questo è ora visto come la” tecnologia di prima generazione ” del sequenziamento del genoma.
Poiché il sequenziamento Sanger (o il metodo di terminazione della catena) è la prima generazione di tecnologia di sequenziamento, comprenderlo è molto importante. Il progetto Genoma umano è iniziato con la tecnologia di sequenziamento Sanger.,
Il metodo di sequenziamento Sanger
Il metodo di sequenziamento Sanger si basa sui dideossinucleotidi (ddNTPs),un tipo di trifosfati deossinucleosidici (dNTPs), che mancano di un gruppo idrossilico 3′ e hanno invece un atomo di idrogeno . Quando queste basi si legano alla crescente sequenza di DNA, terminano la replicazione in quanto non possono legare altre basi. Per eseguire il sequenziamento Sanger, si aggiungono i primer a una soluzione contenente le informazioni genetiche da sequenziare, quindi dividere la soluzione in quattro reazioni PCR., Ogni reazione contiene una miscela con dNTP con uno dei quattro nucleotidi sostituiti con un ddNTP (gruppi ddNTP A, T, G e C). Alla fine della PCR, ciascuna delle quattro reazioni produrrà prodotti PCR di varie lunghezze perché la replica è terminata casualmente. Eseguendo i campioni su un gel con 4 corsie, è possibile mettere insieme la sequenza come ogni sequenza è stata replicata dallo stesso materiale originale. Ecco un esempio in cui i ddNTPs sono in grassetto e i DNTPS non lo sono:
La tua sequenza è ATGCTCAG.,
le quattro reazioni di dare è:
Reazione con ddATP: UNA, ATGCTCA, ATGCTCAG
Reazione con ddGTP: ATG, ATGCTCAG
Reazione con ddCTP: ATGC, ATGCTC, ATGCTCAG
Reazione con ddTTP: A, ATGCT, ATGCTCAG
Tutte le reazioni una volta eseguito un gel sarebbe simile a questa (Immagine da Olwen Reina):
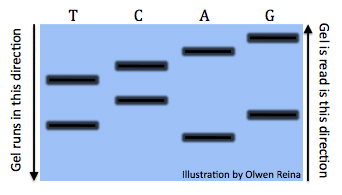
Ogni banda indica le diverse lunghezze di codice. Ad esempio, la band è la destra sotto la” A “simboleggia la sequenza: “ATGCTCA”
Ancora confuso?
Immaginiamo un party game. Il gioco è un gioco di indovinare., Ecco come si gioca:
Stai pensando a un numero e il gruppo deve indovinarlo. La parte difficile è che il numero ha una lunghezza di 200 cifre. Stai leggendo le cifre del numero nella tua testa senza emettere un suono. Ogni tanto una persona ti interrompe e tu dici loro la singola cifra che stavi pensando e dove si trova nella sequenza di 200. Ogni volta che si sono interrotti, si deve ricominciare da capo. Si lascia dopo poche ore e il gruppo deve capire il numero di 200 cifre., Devono mettere insieme le informazioni che hai dato loro, ad esempio il numero 25 era 5, il numero 40 era 0 e così via. Usando le informazioni dalle loro interruzioni, possono ripetere il numero che ti hanno dato.
Anche se questo suona come il gioco più lamest nel mondo, funziona molto bene per il sequenziamento!
Sfortunatamente, è lento, costoso e (in precedenza) si basa su materiali radioattivi. Questo ha spinto gli scienziati a sviluppare nuove e migliori forme di sequenziamento del genoma.,
Next Generation Sequencing Technologies
I più grandi progressi nel sequenziamento del genoma sono stati aumentando la velocità e la precisione, con conseguente riduzione della manodopera e dei costi. La velocità è grazie all’analisi parallela e alla tecnologia ad alta velocità. È la differenza tra ottenere un bambino di 4 anni a leggere Moby Dick e dare un paragrafo ciascuno a un gruppo di major drammatiche e chiedere loro di leggere subito.
Le macchine di sequenziamento sono migliorate selvaggiamente da quando Sanger ha sviluppato il suo metodo., Nel 1987, Applied Biosystems è stato il primo a introdurre una macchina di sequenziamento automatica, chiamata AB370. La macchina si basava su un metodo chiamato elettroforesi capillare che utilizzava il metodo di terminazione della catena di Sanger senza bisogno di un gel.
Alla miscela di reazione PCR vengono aggiunti DDNTP terminanti a catena marcati con fluorescenza. Con il metodo di sequenziamento Sanger, vengono creati prodotti PCR di varie lunghezze e quindi separati in base alle loro dimensioni. La dimensione è misurata dalla carica negativa complessiva del prodotto PCR. Più negativa è la carica, più lungo è il frammento., Prendi l’esempio precedente di incorporazione ddNTP e immagina che le lettere in grassetto siano ddNTPs con un fluorocromo attaccato. Mentre i frammenti vengono tirati verso l’elettrodo positivo di un capillare (vedi immagine sotto), passano un raggio laser che innesca un lampo di luce dal fluorocromo attaccato al ddNTP che è caratteristico del tipo base (ad esempio, verde per A, giallo per T, blu per G, rosso per C). In questo modo, il genoma viene letto attentamente.
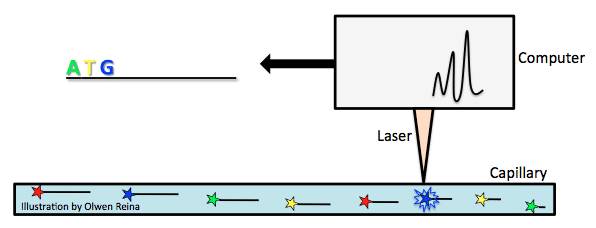
La più grande differenza qui era la velocità e il costo., AB370 poteva rilevare 96 basi contemporaneamente, 500K basi al giorno e aveva una lunghezza di lettura di 600 basi utilizzando un’analisi parallela e una configurazione ad alto throughput. Questa forma di sequenziamento è diventata lo strumento principale per il completamento del progetto genoma umano.
Come si confronta NGS con il sequenziamento di prima generazione?
NGS è caratterizzato da una migliore precisione e velocità, ma anche ridotto manodopera e costi. Non c’è mai stato un momento in cui è stato come a buon mercato, conveniente, o semplice per sequenziare un genoma., Probabilmente, il più grande miglioramento è stato lo sviluppo di analisi parallele, che ha aumentato la velocità di sequenziamento.
L’unica cosa che ci rallenta ora è l’interpretazione dei risultati!

Questo ti ha aiutato? Quindi si prega di condividere con la rete.
















Lascia un commento