Introduction
La réplication dans le noyau des cellules eucaryotes utilise trois ADN polymérases: polymérase α, δ Et ε (Hubscher et al., 2002; Pavlov et coll., 2006b; Kunkel et des Hamburgers, 2008; Loeb et Monnat, 2008; Hamburgers, 2009; Pavlov et Shcherbakova, 2010; Lange et coll., 2011)., La synthèse de L’ADN est directionnelle et passe de 5′ à 3′, où l’attaque nucléophile sur le phosphate α d’un nucléotide par le 3’OH d’une amorce entraîne l’incorporation d’un nucléoside monophosphate et la libération de pyrophosphate (Steitz, 1999). Toutes les ADN polymérases nécessitent une amorce et un 3’OH libre pour effectuer la synthèse de l’ADN, et Pol α ne fait pas exception. Pol α est un hétérotétramère composé de deux sous-unités de primase et de deux sous-unités de polymérase., Les sous-unités de primase initient la réplication de l’ADN en synthétisant des amorces D’ARN courtes (7-12 ribonucléotides), qui sont ensuite prolongées par la polymérase α (Pellegrini, 2012). L’ADN polymérase δ Et ε allongent les amorces générées par Pol α de manière précise et processive (Kunkel, 2004, 2011; Pellegrini, 2012). Chez la levure, L’ADN polymérase δ s’est avérée essentielle pour la synthèse de L’ADN du brin en retard, tandis que pol ε semble fonctionner principalement au niveau du brin principal (Pursell et al., 2007; Nick Mcelhinny et coll., 2008; Kunkel, 2011; Georgescu et coll., 2014)., En revanche, dans les mitochondries, la réplication est la responsabilité d’une seule polymérase, l’ADN polymérase γ (Lee et al., 2009).
Les ADN polymérases sont regroupées en sept familles (A, B, C, D, X, Y et RT). Chez les eucaryotes, les trois ADN polymérases réplicatives nucléaires appartiennent à la famille B (Burgers et al., 2001; Patel et Loeb, 2001). Il existe maintenant des structures cristallines des trois ADN polymérases réplicatives de levure, qui permettent pour la première fois une comparaison de leurs éléments structurels communs ainsi qu’une étude de leurs caractéristiques uniques (Swan et al., 2009; Perera et coll.,, 2013; Hogg et coll., 2014; Jain et coll., 2014a). Les trois ADN polymérases réplicatives sont des enzymes multi-sous-unités (Tableau 1) (Johansson et Macneill, 2010; Pavlov et Shcherbakova, 2010; Makarova et al., 2014). L’objectif principal de cette revue est leur domaine catalytique, ou sous-unité A.
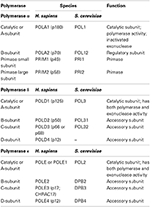
Tableau 1. Les ADN polymérases eucaryotes sont des enzymes multi-sous-unités.,
structure globale des polymérases de la famille B
toutes les ADN polymérases partagent un pli polymérase commun, qui a été comparé à une main droite humaine, composé de trois sous-domaines: les doigts, la paume et le pouce (Steitz, 1999; Patel et Loeb, 2001). La paume, un pli hautement conservé composé de quatre brins β antiparallèles et de deux hélices, abrite deux aspartates catalytiques strictement conservés situés dans le motif A, DXXLYPS et le motif C, DTDS (Delarue et al., 1990; Braithwaite et Ito, 1993)., Ce pli de type RRM est partagé par un très grand groupe d’enzymes, y compris L’ADN et L’ARN polymérases, les transcriptases inverses, la polymérase CRISPR et même les transférases inverses (3′-5′) telles que le Thg1 (Anantharaman et al., 2010; Hyde et coll., 2010). En revanche, les sous-domaines du pouce et des doigts présentent une diversité structurelle beaucoup plus grande (Steitz, 1999). Les doigts subissent un changement conformationnel lors de la liaison de l’ADN et du nucléotide entrant correct. Ce mouvement permet aux résidus dans le sous-domaine des doigts d’entrer en contact avec le nucléotide dans la paire de bases naissante., Le pouce maintient le duplex D’ADN pendant la réplication et joue un rôle dans la processivité (Doublié et Ellenberger, 1998; Doublié et al., 1999).
Les ADN polymérases eucaryotes α, δ Et ε partagent une homologie avec de nombreuses polymérases archéales, bactériennes, bactériophages et virales (Delarue et al., 1990; Braithwaite et Ito, 1993; Franklin et coll., 2001; Firbank et coll., 2008; Wang et Yang, 2009)., Koonin et ses collaborateurs ont fourni une analyse phylogénétique détaillée des ADN polymérases archéales et de leur relation avec les polymérases eucaryotes dans ce numéro de Frontiers in Microbiology dédié aux polymérases Makarova et al. (2014).
toutes les polymérases de la famille B sont composées de cinq sous-domaines: les doigts, le pouce et la paume (décrits ci-dessus) constituant le cœur de l’enzyme, ainsi qu’un domaine exonucléase et un domaine N-terminal (NTD) (Franklin et al., 2001; Xia et Konigsberg, 2014) (Figure 1; Tableau S1)., Le domaine exonucléase porte une activité de relecture 3′-5′, qui élimine les nucléotides mal incorporés. Le site actif de l’exonucléase est situé à 40-45 Å du site actif de la polymérase. La MTN semble être dépourvue d’activité catalytique. Dans Pol δ, la NTD comprend trois motifs: l’un a une topologie ressemblant à un pli OB, un motif de liaison à L’ADN monocaténaire, et un autre porte un motif de liaison à L’ARN (Motif de reconnaissance de L’ARN ou RRM) (Swan et al., 2009). Chez le bactériophage T4, Les mutations de la MTN diminuent l’expression de la polymérase (Hughes et al., 1987)., Dans RB69 et T4, la polymérase gp43 lie son propre ARN messager, vraisemblablement à travers la NTD et réprime la traduction (Petrov et al., 2002), ce qui ne semble pas être le cas de pol δ (Swan et coll., 2009). De nouvelles données indiquent que les MTN jouent un rôle dans la stabilité et la fidélité de la polymérase par ses interactions avec d’autres domaines (Li et al., 2010; Prindle et coll., 2013) (voir ci-dessous).
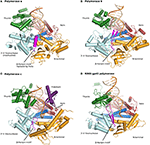
la Figure 1. Les complexes ternaires des polymérases α, δ, ε et rb69 gp43 sont illustrés à partir d’orientations identiques à des fins de comparaison., Les domaines du pouce (vert) et des doigts (bleu foncé) saisissent l’acide nucléique duplex (amorce montrée en beige, gabarit en orange) contre le domaine de la paume (rouge). Le domaine n-terminal apparaît dans l’or, adjacent au domaine exonucléase 3′ -5 ‘ (cyan). (A) la polymérase α (PDBID 4FYD) lie un hybride ARN/ADN, où le sillon mineur large et peu profond de L’ADN de forme A est apparent près du pouce. Le domaine exonucléase 3 ‘ -5 ‘ est dépourvu d’activité. Une région hélicoïdale (magenta) dans le domaine exonucléase inactivé stabilise l’extrémité 5 du gabarit., (B) la polymérase δ (PDBID 3IAY) présente un grand motif β En Épingle À Cheveux (magenta), ce qui est important pour faire passer le brin d’amorce du site actif de la polymérase au site actif de l’exonucléase en cas de relecture. (C) la polymérase ε (PDBID 4M8O) possède un domaine P unique (violet), qui confère à la polymérase une processivité accrue. Fait intéressant, le motif β En Épingle À Cheveux est atrophié dans Pol ε., (D) la Conservation du pli de L’ADN polymérase de la famille B et l’organisation du domaine sont évidentes lorsque l’enzyme modèle du bactériophage RB69 gp43 (PDBID 2OZS) est observée avec les trois polymérases réplicatives eucaryotes. La délimitation du domaine pour chaque polymérase est donnée dans le tableau S1. Figure a été faite avec PyMOL (le système graphique moléculaire PyMOL, version 1.5.0.4 Schrödinger, LLC.).
toutes les ADN polymérases de la famille B des mammifères sont connues pour abriter deux sites de liaison aux métaux riches en cystéine (CysA et CysB) dans leur domaine C-terminal (CTD) (Figure 2)., CysA est présumé être un site de liaison au zinc alors que CysB est un cluster fer soufre (Netz et al., 2012). La perte de la grappe dans le CTD de la levure pol δ affecte négativement les interactions avec sa sous-unité B accessoire (Sanchez Garcia et al., 2004). Le motif de liaison au zinc s’est avéré important pour l’interaction de pol δ Avec son facteur de processivité, PCNA (Netz et al., 2012).

la Figure 2. Diagramme schématique des trois Saccharomyces cerevisiae ADN polymérases réplicatives α, δ Et ε., L’ADN polymérase du bactériophage RB69 est présentée à titre de comparaison.
l’ADN Polymérase α
La sous-unité catalytique de l’ADN polymérase α est composé de 1468 acides aminés (Tableau 2). La structure protéique conçue pour la cristallisation a été tronquée aux termini N et C (résidus 349-1258) et n’a donc pas le CTD et son cluster (Figure 2). La construction a été cristallisée sans ligand, dans un complexe binaire avec un oligonucléotide hybride ADN / ARN, et dans un complexe ternaire avec ADN/ARN et nucléotide entrant (Perera et al., 2013) (Figure 1).,

le Tableau 2. Compilation D’ADN polymérases de la famille B de structure connue.
Un Mécanisme de Désengagement de la Polymérase
L’ARN/ADN oligonucléotide capturé dans les cristaux adopte Une forme de conformation, comme prévu. Le domaine du pouce s’engage dans de multiples interactions avec l’amorce D’ARN, à la fois via des contacts hydrophobes et des interactions polaires (Perera et al., 2013)., Des expériences en solution ont montré que l’extension de l’amorce D’ARN par Pol α est limitée à 10-12 nucléotides, ce qui équivaut à un tour d’hélice. Cette observation a conduit les auteurs à suggérer un mécanisme de terminaison de la synthèse d’amorces par Pol α dans lequel la perte d’interactions spécifiques entre le pouce et l’ARN déclencherait le désengagement de la polymérase de l’oligonucléotide ADN/ARN, et permettrait une main sur une polymérase réplicative.,
les mouvements dans le domaine palmier peuvent faciliter la Translocation de Pol α
la cristallisation de l’enzyme dans trois états (apo, binaire et ternaire) a permis aux auteurs de superposer les trois modèles structuraux. Pol α est la seule ADN polymérase de la famille eucaryote B pour laquelle les trois états ont été capturés dans une structure cristalline. La superposition structurelle a révélé qu’en plus des mouvements bien documentés des sous-domaines des doigts et du Pouce accompagnant la liaison au substrat et le transfert nucléotidyle, le sous-domaine de la paume lui-même subit un réarrangement structurel (Perera et al.,, 2013). Les auteurs proposent que les différentes conformations du domaine palm pourraient faciliter la translocation de Pol α le long et au-delà du duplex ARN/ADN. Comme mentionné ci-dessus, la perte de contacts avec le brin D’ARN est prévue pour déclencher la libération de l’amorce, qui devient alors disponible pour l’extension par Pol δ OU ε.,
un repli protéique différent dans le sous-domaine exonucléase inactivé
l’activité de relecture est abolie dans Pol α, En raison de mutations dans les quatre carboxylates (Asp114/Glu116/Asp222/Asp327 dans RB69 gp43 correspondent à Ser542/Gln544/Tyr644/Asn757 dans un alignement basé sur la structure) (Tableau 2). De plus, le motif β-épingle à cheveux trouvé dans la plupart des polymérases de la famille B (résidus 246-267 dans RB69 gp43) est remplacé par une région hélicoïdale dans Pol α (résidus 667-676; 681-693) (Hogg et al., 2007)., L’épingle β fait partie du domaine exonucléase et il a été démontré dans les pols T4 et RB69 qu’elle participe au partage de l’amorce D’ADN entre la polymérase et le site actif exonucléase (Reha-Krantz, 1988; Stocki et al., 1995; Hogg et coll., 2007). En l’absence d’activité de relecture, il n’est pas surprenant que ce motif n’ait pas été retenu dans pol α. Résidus His 684 et Phe 685 de la région hélicoïdale dans l’empilement Pol α Avec une base thymine et guanine, respectivement, aux positions -3 et -2 dans le 5 ‘ end non apparié du gabarit (Perera et al., 2013)., Ainsi, dans pol α, la région correspondant au motif β-épingle à cheveux adopte un pli différent (hélices vs brins β) et une fonction différente (stabilisation de la région non appariée du brin de gabarit plutôt que de faciliter la commutation de site actif). Puisque Pol α est dépourvu d’activité de relecture, la question se pose de savoir si les oligonucléotides courts sont corrigés, et si oui, par quel ADN polymérase. Il semble que la relecture des amorces synthétisées par pol α soit effectuée par pol δ (Pavlov et al., 2006a).,
ADN polymérase δ
Le Pol δ humain est composé de quatre sous-unités alors que Saccharomyces cerevisiae en a trois (Gerik et al., 1998; Liu et coll., 2000) (Tableau 1). En plus de sa fonction dans la réplication de L’ADN, Pol δ joue un rôle dans la réparation et la recombinaison de l’ADN (Hubscher et al., 2002; Lee et coll., 2012; Tahirov, 2012). P12, la plus petite sous-unité chez l’homme Pol δ et aussi la sous-unité qui n’est pas observée chez la levure en herbe, est dégradée en réponse à des dommages à l’ADN (Lee et al., 2014). La sous-unité catalytique de la levure Pol δ (POL3) est composée de 1097 résidus., La construction utilisée pour la cristallisation comprend les résidus 67-985 et n’a donc pas le CTD (Figure 1; Tableau 2).
un troisième Ion métallique dans le Site actif de la polymérase
le domaine palm contient trois carboxylates conservés (Asp608, Asp762 et Asp764). Les deux aspartates catalytiques, Asp608 et Asp764, entrent en contact avec deux ions métalliques (Ca2+) dans le site actif de la polymérase séparés par 3,7 Å. Curieusement, un troisième métal a été observé coordonné par le phosphate γ du nucléotide entrant et Glu802, avec Glu800 à proximité., La mutation des deux glutamates en alanine a donné une variante de la polymérase avec une efficacité d’incorporation réduite pour les nucléotides corrects et incorrects (Swan et al., 2009). À ces positions d’acides aminés, pol α et pol ε ont également des résidus carboxylates (pol δ Glu800/Glu802 correspondent à pol α Asp1033/Asp1035 et pol ε Glu945/Asp947). Il reste à déterminer si ces carboxylates jouent des rôles similaires dans Pol α Et ε.,
haute fidélité et relecture
La pol δ humaine est une polymérase haute fidélité, catalysant la réaction de transfert nucléotidyle avec une fréquence d’erreur de 1 pour 22 000 (Schmitt et al., 2009). La relecture augmente la fidélité de la polymérase d’un facteur 10-100 (Mcculloch et Kunkel, 2008; Prindle et al., 2013). Pol δ abrite un site actif de polymérase et d’exonucléase, séparé d’environ 45 Å (Swan et al., 2009). Les ADN polymérases ayant une activité de relecture sont capables de détecter des nucléotides mal incorporés en entrant en contact avec le sillon mineur des paires de bases au-delà du site d’insertion., La protéine interagit avec les accepteurs universels de liaisons hydrogène aux positions N3 et O2 des purines et des pyrimidines, respectivement (Seeman et al., 1976; Doublié et coll., 1998; Franklin et coll., 2001). Ces contacts de liaison hydrogène sont préservés lorsque la paire de bases adopte une géométrie Watson-Crick et perdus en cas de non-concordance. Dans rb69 gp43, les contacts s’étendent aux deux premières paires de bases au-delà de la paire de bases naissante (Franklin et al., 2001; Hogg et coll., 2004, 2005). Les contacts sont beaucoup plus étendus dans Pol δ, s’étendant à cinq paires de bases post-insertion (Swan et al.,, 2009), ce qui pourrait contribuer à sa haute fidélité.
comme mentionné ci-dessus, le segment β-hairpin du domaine exonucléase joue un rôle critique dans la partition de l’ADN entre les sites de polymérisation et de relecture dans les pols T4 et RB69 (Stocki et al., 1995; Hogg et coll., 2007). Dans RB69 gp43, le motif en épingle à cheveux β adopte différentes conformations, selon que le complexe a été obtenu avec de l’ADN intact (Franklin et al., 2001; Zahn et coll., 2007) ou de L’ADN contenant un dommage (Freisinger et al., 2004; Hogg et coll., 2004)., Il a été entièrement visualisé en contactant à la fois l’amorce et les brins de gabarit dans un complexe avec du thymine glycol (Aller et al., 2011). De même, l’épingle β dans pol δ fait saillie dans la rainure principale de l’ADN et agit comme un coin entre L’ADN double brin et le brin 5 ‘ end simple brin du brin gabarit, qui est stabilisé par deux résidus aromatiques Phe441 et Tyr446 (Figure 1) (Swan et al., 2009). La position de l’épingle β est compatible avec un rôle dans le changement de site actif.,
contacts Interdomains et fidélité
Les Mutations impliquées dans le cancer se trouvent principalement dans le domaine exonucléase de Pol δ Et ε, soulignant le rôle critique de la relecture dans la réduction de l’incidence des mutations (Church et al., 2013; Henninger et Pursell, 2014). Une mutation dans les cellules cancéreuses colorectales humaines se localise au domaine des doigts, R689W. la mutation analogue dans la levure (R696W) entraîne un phénotype mutateur (Daee et al., 2010)., Une mutation au voisinage de Arg696 dans le motif B hautement conservé du sous-domaine fingers de la levure Pol δ (A699Q) entraîne également un phénotype mutateur. Cette région des doigts est à proximité de la MTN. La mutation Met540 de la NTD en alanine abolit le phénotype mutateur de A699Q, illustrant que les interactions entre les doigts et la NTD peuvent affecter la fidélité de la polymérase (Prindle et al., 2013)., De même, dans les pols T4 et RB69, le motif du noyau NPL, qui implique des résidus des domaines N-terminal et palm, est en contact avec le domaine fingers et a été montré pour stabiliser les complexes polymérase-ADN (Li et al., 2010).
ADN polymérase ε
la sous-unité catalytique de L’ADN polymérase ε est le produit d’un très grand gène (2222 acides aminés chez la levure; 2286 chez l’homme), et n’est que troisième en taille après la polymérase ζ (également membre de la famille B) et pol θ, une polymérase de la famille A (3130 et 2590 acides aminés, respectivement, chez l’homme) (Lange et al.,, 2011; Hogg et Johansson, 2012) (Figure 1; Tableau 2). Pol ε est deux fois plus grand que pol δ et est composé de deux régions de polymérase/exonucléase en tandem. Le segment N-terminal possède à la fois des activités de polymérase et de relecture, tandis que le segment C-terminal est inactivé. Les deux modules exonucléase-polymérase sont distants (Tahirov et al., 2009). Bien que l’on présume que le segment inactivé joue un rôle structurel lors de la réplication, deux groupes ont pu cristalliser des constructions Pol ε catalytiquement actives (résidus 1-1228; 1-1187) dépourvues de tout le module C-terminal (Hogg et al.,, 2014; Jain et coll., 2014a). Les deux structures cristallines étaient constituées d’un complexe ternaire de la polymérase, d’une amorce/matrice D’ADN et d’un nucléotide entrant.
un nouveau domaine de Processivité émanant du domaine Palm
Pol ε diffère de pol δ En ce qu’il ne nécessite pas la pince coulissante PCNA pour une processivité élevée (Hogg et Johansson, 2012). Le domaine palm de pol ε est sensiblement plus grand (380 résidus) que celui de pol α OU δ (175 et 203 résidus, respectivement)., Les récentes structures cristallines Pol ε ont révélé que les insertions dans le domaine palm forment collectivement un nouveau domaine composé de trois brins β et de deux hélices (résidus 533-555; 682-760) (Hogg et al., 2014; Jain et coll., 2014a) (Figure 1; Tableau S1). La suppression des résidus 690-751 a entraîné une variante avec une diminution de l’activité de la polymérase. De plus, les résidus chargés positivement en mutation (His748, Arg749 et Lys751) situés à proximité de l’épine dorsale phosphatée ont affecté la processivité de l’enzyme (Hogg et al., 2014)., Le domaine supplémentaire provenant de la paume a ainsi été nommé processivité ou domaine P, d’après sa fonction. La base du domaine P abrite un site de liaison métallique (voir ci-dessous) (Hogg et al., 2014; Jain et coll., 2014a, b).
une grappe de fer soufre dans le domaine de la polymérase
des études inattendues en solution ont révélé que la sous-unité catalytique de la polymérase ε de levure contient elle-même une grappe dans son pli de polymérase (Jain et al., 2014b), en plus du groupe dans le CTD (Figure 2; Tableau 2)., Le deuxième groupe au sein de pol ε suggère que cette polymérase peut être plus sensible au stress oxydatif (Jain et al., 2014b). Les structures cristallines de pol ε, cependant, n’ont pas révélé d’amas dans le domaine de la polymérase (Hogg et al., 2014; Jain et coll., 2014a; Zahn et Doublié, 2014). Deux des résidus de cystéine sont désordonnés dans les modèles structuraux et le site de liaison métallique qui en résulte semble lier le zinc (Hogg et al., 2014; Jain et coll., 2014a). La Substitution de A par un zinc non natif dans les protéines de liaison aux métaux n’est pas inhabituelle (Netz et al., 2012) car les grappes sont labiles., La visualisation de l’intérieur du domaine polymérase de pol ε peut nécessiter des conditions anaérobies.
un court motif en épingle à cheveux β dans le domaine exonucléase
dans toute ADN polymérase abritant à la fois des activités de polymérase et d’exonucléase, L’ADN lié est en équilibre entre les deux centres actifs (Beechem et al., 1998). La concentration de nucléotide entrant et la présence d’une base endommagée ou d’une mauvaise paire sont deux facteurs qui influencent le transfert de L’ADN du site d’activation de la polymérase vers le site actif de relecture., Les polymérases surveillent le côté mineur de la rainure des paires de bases nouvellement formées et interagissent avec les accepteurs universels de Liaison H, O3 et N2, comme moyen de vérifier les inadéquations (Seeman et al., 1976; Franklin et coll., 2001). Une caractéristique unique de pol ε est le contact avec le côté principal de la rainure de la paire de bases naissante via un résidu du domaine exonucléase, Tyr431. Une analyse plus approfondie est nécessaire pour élucider le rôle potentiel de cette tyrosine dans la haute fidélité de pol ε.,
dans Pol δ, le segment β-épingle à cheveux s’insère dans l’ADN et agit comme un coin entre L’ADN simple brin et l’ADN double brin (Swan et al., 2009). Dans L’ADN pol II D’E. coli, l’insertion d’un barillet β déplace la position de l’épingle β de telle sorte que la polymérisation est favorisée par rapport à la relecture (Wang et Yang, 2009). Cette modification permet vraisemblablement à cette polymérase de réaliser une extension de synthèse de translation., Puisque pol ε est une ADN polymérase précise, l’hypothèse avant la connaissance de la structure cristalline serait que l’épingle β devrait être plus proche de celle de pol δ Que de celle de E. coli Pol II. étonnamment, le motif de l’épingle β dans pol ε est tronqué, trop court pour entrer en contact avec L’ADN (Figure 1). Quel motif protéique, alors, pourrait faciliter la commutation de site actif lors de la détection d’une mauvaise paire? Le domaine P est un bon candidat, en raison de ses contacts avec les brins d’amorce et de modèle; les résidus du domaine P pourraient détecter des erreurs de réplication et ainsi faciliter la commutation de site active.,
Conclusions
Les Trois ADN polymérases réplicatives eucaryotes utilisent un pli commun de la famille B, et chaque polymérase a incorporé des éléments structuraux modifiés qui sont uniques et adaptés à la fonction spécifique de chaque polymérase (par exemple, l’ajout du domaine de processivité dans pol ε, une polymérase processive qui n’utilise pas de PCNA, ou la région modifiée entrant en contact avec le 5 ‘ end du gabarit dans pol α, une polymérase dépourvue d’activité de relecture). Le pli des polymérases de la famille B est bien adapté aux polymérases réplicatives haute fidélité., Mais étonnamment, il est également utilisé par les polymérases de translation. Eukaryotic pol ζ (OU REV3L) est une polymérase de 353 kDa qui fonctionne dans la synthèse de translation et semble supprimer la tumorigenèse (Wittschieben et al., 2010; Lange et coll., 2011; Zahn et coll., 2011; Hogg et Johansson, 2012; Sharma et coll., 2013). La structure D’E. coli Pol II a révélé des modifications dans les NTD qui affectent la position de l’épingle β du domaine exonucléase, et donc le cloisonnement de l’ADN entre les sites de polymérisation et de relecture (Wang et Yang, 2009)., La structure de Pol ζ peut révéler des ajustements similaires, qui modifient le pli employé par les polymérases réplicatives haute fidélité pour rendre l’enzyme moins fidèle et capable d’effectuer la synthèse de translation.
déclaration de conflit d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière pouvant être interprétée comme un conflit d’intérêts potentiel.
reconnaissance
Ce travail a été soutenu par une subvention des National Institutes of Health (NCI R01 CA 52040).
















Laisser un commentaire