l’achèvement du Projet Génome Humain en 2003 a inauguré une nouvelle ère d’analyse rapide, abordable et précise du génome—appelée Next Generation Sequencing (NGS). NGS s’appuie sur les technologies de” séquençage de première génération » pour produire des résultats de séquençage précis et rentables.
Fred Sanger a séquencé le premier génome D’ADN entier, le virus phage ?X174, en 1977., La même année, Sanger a développé la future épine dorsale de l’ère du génome: la technologie de séquençage de l’ADN. Sa technique a utilisé la méthode de terminaison de chaîne. Ceci est maintenant considéré comme la” technologie de première génération » du séquençage du génome.
étant donné que le séquençage Sanger (ou la méthode de terminaison de chaîne) est la première génération de technologie de séquençage, il est très important de la comprendre. Le Projet Génome Humain a commencé avec la technologie de séquençage Sanger.,
la méthode de séquençage de Sanger
la méthode de séquençage de Sanger repose sur des didésoxynucléotides (ddNTPs),un type de désoxynucléoside triphosphates (dNTPs), qui n’ont pas de groupe hydroxyle 3′ et ont un atome d’hydrogène à la place . Lorsque ces bases se lient à la séquence D’ADN en croissance, elles mettent fin à la réplication car elles ne peuvent pas lier d’autres bases. Pour effectuer le séquençage Sanger, vous ajoutez vos amorces à une solution contenant l’information génétique à séquencer, puis divisez la solution en quatre réactions PCR., Chaque réaction contient un mélange avec dNTP avec l’un des quatre nucléotides substitués par un ddNTP (groupes A, T, G et C ddNTP). À la fin de la PCR, chacune de vos quatre réactions donnera des produits PCR de différentes longueurs car la réplication est terminée de manière aléatoire. En exécutant les échantillons sur un gel à 4 voies, vous pouvez assembler la séquence car chaque séquence a été répliquée à partir du même matériau d’origine. Voici un exemple où les ddNTPs sont en gras et les dNTPs ne le sont pas:
votre séquence est ATGCTCAG.,
Vos quatre réactions vous donnent:
réaction avec ddATP: A, ATGCTCA, ATGCTCAG
réaction avec ddGTP: ATG, ATGCTCAG
réaction avec ddCTP: ATGC, ATGCTC, ATGCTCAG
réaction avec ddTTP: AT, ATGCT, ATGCTCAG
toutes les réactions une fois exécuté un gel ressemblerait à ceci (Image par Olwen Reina):
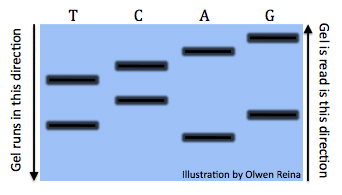
chaque bande indique le code de longueurs différentes. Par exemple, la bande est la droite sous le « A” symbolise la séquence: « ATGCTCA »
toujours confus?
imaginons un jeu de société. Le jeu est un jeu de devinettes., Voici comment il est joué:
Vous pensez à un nombre et le groupe a de le deviner. La partie délicate est que le nombre est de 200 chiffres. Vous lisez les chiffres du nombre dans votre tête sans faire de bruit. De temps en temps, une personne vous interrompt et vous lui dites le seul chiffre que vous pensiez et où il se trouve dans la séquence de 200. Chaque fois que vous êtes interrompu, vous devez recommencer. Vous partez après quelques heures et le groupe doit trouver le numéro à 200 chiffres., Ils doivent rassembler les informations que vous leur avez données, par exemple le 25e numéro était 5, le 40e numéro était 0, etc. En utilisant les informations de leurs interruptions, ils peuvent répéter le numéro qu’ils vous ont donné.
bien que cela ressemble au jeu le plus difficile au monde, il fonctionne très bien pour le séquençage!
malheureusement, il est lent, coûteux et (auparavant) repose sur des matières radioactives. Cela a poussé les scientifiques à développer de nouvelles et meilleures formes de séquençage du génome.,
technologies de séquençage de nouvelle génération
Les plus grandes avancées dans le séquençage du génome ont été l’augmentation de la vitesse et de la précision, entraînant une réduction de la main-d’œuvre et des coûts. La vitesse est grâce à l’analyse parallèle et à la technologie à haut débit. C’est la différence entre faire lire Moby Dick à un enfant de 4 ans et donner un paragraphe à un groupe de majors du théâtre et leur demander de lire à la fois.
Les machines de séquençage se sont considérablement améliorées depuis que Sanger a développé sa méthode., En 1987, Applied Biosystems est devenu le premier à introduire une machine de séquençage automatique, appelée AB370. La machine reposait sur une méthode appelée électrophorèse capillaire qui utilisait la méthode de terminaison de chaîne de Sanger sans avoir besoin de gel.
des ddNTP à terminaison de chaîne marqués Fluorescemment sont ajoutés au mélange réactionnel de PCR. Par la méthode de séquençage Sanger, des produits PCR de différentes longueurs sont créés, puis séparés en fonction de leur taille. La taille est mesurée par la charge négative globale du produit PCR. Plus la charge est négative, plus le fragment est long., Prenez l’exemple ci-dessus de l’incorporation ddNTP et imaginez que les lettres en gras sont ddNTPs avec un fluorochrome attaché. Comme les fragments sont tirés vers l’électrode positive d’un capillaire (voir image ci-dessous), ils passent un faisceau laser qui déclenche un flash de lumière du fluorochrome attaché au ddNTP qui est caractéristique du type de base (par exemple, vert pour A, jaune pour T, bleu pour G, rouge pour C). De cette façon, le génome est lu attentivement.
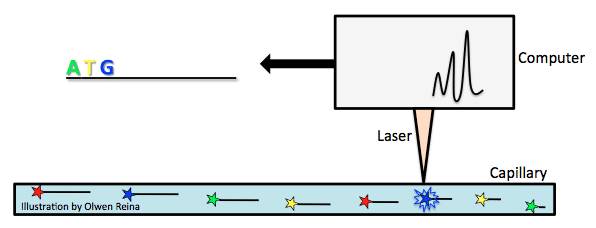
La plus grande différence ici, c’était la vitesse et de coût., AB370 pouvait détecter 96 bases à la fois, 500K bases par jour, et avait une longueur de lecture de 600 bases en utilisant une analyse parallèle et une configuration à haut débit. Cette forme de séquençage est devenue l’outil principal pour l’achèvement du projet Génome Humain.
comment NGS se Compare-t-il au séquençage de première génération?
NGS se caractérise par une précision et une vitesse améliorées, mais également une réduction de la main-d’œuvre et des coûts. Il n’y a jamais eu un moment où il a été aussi bon marché, pratique ou simple de séquencer un génome., Sans doute, la plus grande amélioration a été le développement de l’analyse parallèle, qui a augmenté la vitesse de séquençage.
la seule chose qui nous ralentit maintenant est l’interprétation des résultats!

A cette vous a aidé? Ensuite, veuillez partager avec votre réseau.
















Laisser un commentaire