Introduction
Replication in the nucleus of eukaryotic cells employs three DNA polymerases: polymerase α, δ ,And ε (Hubscher et al., 2002; Pavlov et al., 2006b; Kunkel and Burgers, 2008; Loeb and Monnat, 2008; Burgers, 2009; Pavlov and Shcherbakova, 2010; Lange et al., 2011)., A síntese do ADN é direccional e procede de 5 ‘a 3’, onde o ataque nucleofílico ao fosfato α de um nucleótido pelo 3’OH de um iniciador resulta na incorporação de um monofosfato de nucleósido e na libertação de pirofosfato (Steitz, 1999). Todas as polimerases de ADN requerem um iniciador e um 3’Oh livre para conduzir a síntese de ADN, e o pol α Não é excepção. Pol α é um heterotetramer composto por duas subunidades de primase e duas subunidades de polimerase., As subunidades da primase iniciam replicação de DNA sintetizando primers curtos (7-12 ribonucleotídeos) de RNA, que são então estendidos pela polimerase α (Pellegrini, 2012). DNA polimerase δ And ε elongate the primers generated by pol α in an accurate and processive manner (Kunkel, 2004, 2011; Pellegrini, 2012). Na levedura, demonstrou-se que a ADN polimerase δ É essencial para a síntese de ADN da cadeia atrasada, enquanto que a pol ε parece funcionar principalmente na cadeia líder (Pursell et al., 2007; Nick Mcelhinny et al., 2008; Kunkel, 2011; Georgescu et al., 2014)., Em contraste, na replicação mitocôndria é a responsabilidade de uma única polimerase, DNA polimerase γ (Lee et al., 2009).
As polimerases do ADN estão agrupadas em sete famílias (a, B, C, D, X, Y e RT). Nas eucariotas, as três polimerases do ADN replicativo nuclear pertencem, por acaso, à família B (Burgers et al., 2001; Patel e Loeb, 2001). Existem agora estruturas de cristais de todas as três polimerases replicativas do DNA a partir de leveduras, que permitem pela primeira vez uma comparação de seus elementos estruturais compartilhados, bem como um estudo de suas características únicas (Swan et al., 2009; Perera et al.,, 2013; Hogg et al., 2014; Jain et al., 2014a). As três polimerases replicativas do ADN são enzimas multi-subunidades (Tabela 1) (Johansson e Macneill, 2010; Pavlov e Shcherbakova, 2010; Makarova et al., 2014). O foco principal desta revisão está em seu domínio catalítico, ou subunidade A.
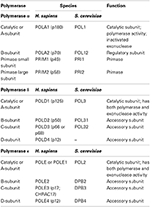
Tabela 1. As polimerases do ADN eucarióticas são enzimas multi-subunitárias.,
estrutura global das polimerases da família B
Todas as polimerases do ADN partilham uma dobra de polimerase comum, que foi comparada a uma mão direita humana, composta por três subdomínios: dedos, palma e polegar (Steitz, 1999; Patel e Loebe, 2001). A palma, uma dobra altamente conservada composta de quatro cordões β antiparalelas e duas hélices, abriga dois aspartatos catalíticos rigorosamente conservados localizados em motif a, DXXLYPS e motif C, DTDS (Delarue et al., 1990; Braithwaite and Ito, 1993)., Este MRR-como dobrar é compartilhado por um grupo muito grande de enzimas, incluindo DNA e RNA polimerases, inversa transcriptases, CRISPR polimerase, e até mesmo reverter (3′-5′) transferases, tais como Thg1 (Anantharaman et al., 2010; Hyde et al., 2010). Em contraste, os subdomínios do polegar e dos dedos exibem substancialmente mais diversidade estrutural (Steitz, 1999). Os dedos sofrem uma mudança conformacional ao ligarem o DNA e o nucleótido de entrada correto. Este movimento permite que resíduos no subdomínio dos dedos entrem em contato com o nucleótido no par base nascente., The thumb holds the DNA duplex during replication and plays a part in processivity (Doublié and Ellenberger, 1998; Doublié et al., 1999).
eucariotic DNA polimerases α, δ, And ε share homology with many archaeal, bacteriophage, and viral polymerases (Delarue et al., 1990; Braithwaite and Ito, 1993; Franklin et al., 2001; Firbank et al., 2008; Wang and Yang, 2009)., Koonin e colaboradores contribuíram com uma detalhada análise filogenética de bactérias DNA polimerases e a sua relação com eucarióticas polimerases neste problema de Fronteiras em Microbiologia dedicado a polimerases Makarova et al. (2014).
Todas as polimerases da família B são compostas por cinco subdomínios: os dedos, polegar e Palma (descritos acima) que constituem o núcleo da enzima, bem como um domínio de exonuclease e um domínio N-terminal (NTD) (Franklin et al., 2001; Xia e Konigsberg, 2014) (Figura 1; Quadro S1)., O domínio da exonuclease transporta uma actividade de revisão de 3′-5′, que remove nucleótidos misincorporados. O local activo da exonuclease está localizado a 40-45 Å do local activo da polimerase. A DNT parece ser desprovida de actividade catalítica. In pol δ the NTD comprises three motifs: one has a topology resembling an OB fold, a single-stranded DNA binding motif, and another bears an RNA-binding motif (RNA Recognition Motif or RRM) (Swan et al., 2009). No bacteriófago T4, as mutações na DMT diminuem a expressão da polimerase (Hughes et al., 1987)., Na RB69 e T4, a polimerase gp43 liga seu próprio RNA mensageiro, presumivelmente através da NTD e reprime a tradução (Petrov et al., 2002), o que não parece ser o caso de pol δ (Swan et al., 2009). Novos dados indicam que a DNT desempenha um papel na estabilidade e fidelidade da polimerase através de suas interações com outros domínios (Li et al., 2010; Prindle et al., 2013) (ver abaixo).
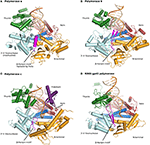
a Figura 1. Complexos ternários de polimerases α, δ, ε e RB69 gp43 são ilustrados a partir de orientações idênticas para comparação., O polegar (verde) e os dedos (azul escuro) agarram o ácido nucleico duplex (primer mostrado em bege, modelo em laranja) contra o domínio da palma (vermelho). O domínio N-terminal aparece em ouro, adjacente ao Domínio 3′ -5 ‘ exonuclease (cyan). A) a polimerase α (PDBID 4FYD) liga-se a um híbrido RNA/ADN, onde a ranhura menor larga e superficial do ADN da forma A é aparente perto do polegar. O domínio da exonuclease 3 ‘ -5 ‘ é desprovido de actividade. Uma região helicoidal (magenta) no domínio das exonucleases inactivadas estabiliza a extremidade 5 do modelo., B) a polimerase δ (PDBID 3IAY) abriga um grande motivo de alfinete β (magenta), que é importante para mudar a linha de iniciação do local activo da polimerase para o local activo da exonuclease em caso de revisão. C) a polimerase ε (PDBID 4M8O) possui um domínio P único (púrpura), que confere à polimerase maior processividade. Curiosamente, o motivo do alfinete β é atrofiado em Pol ε., (D) A conservação da dobra de ADN polimerase da família B, e a organização do domínio, é evidente quando a enzima modelo da bacteriofagem RB69 gp43 (PDBID 2OZS) é vista juntamente com as três polimerases replicativas eucarióticas. A delimitação de domínio para cada polimerase é dada na tabela S1. Figura foi feita com PyMOL (o sistema gráfico Molecular PyMOL, versão 1.5.0.4 Schrödinger, LLC.).
todas as polimerases de ADN da família B de mamíferos são conhecidas por abrigarem dois locais de ligação de Metais ricos em cisteína (CysA e CysB) no seu domínio C-terminal (CTD) (Figura 2)., CysA é presumivelmente um local de ligação ao zinco, enquanto CysB é um aglomerado de enxofre de ferro (Netz et al., 2012). A perda do aglomerado no CTD da levedura pol δ afeta negativamente as interações com sua subunidade B acessória (Sanchez Garcia et al., 2004). O motivo de ligação ao zinco demonstrou ser importante para a interacção de pol δ Com o seu Factor de processividade, PCNA (Netz et al., 2012).

Figura 2. Diagrama esquemático das três polimerases ADN replicativas da Saccharomyces cerevisiae α, δ e ε., A DNA polimerase de bacteriophage RB69 é mostrada para comparação.
ADN polimerase α
a subunidade catalítica da ADN polimerase α é composta por 1468 aminoácidos (Quadro 2). A construção proteica projetada para cristalização foi truncada no n – E C-termini (resíduos 349-1258) e, portanto, falta o CTD e seu aglomerado (Figura 2). A construção não foi cristalizada, em um complexo binário com um oligonucleótido híbrido DNA / RNA, e em um complexo ternário com DNA/RNA e nucleótido recebido (Perera et al., 2013) (Figura 1).,

Tabela 2. Compilation of DNA polymerases of the B family of known structure.
um mecanismo de Desengajamento da polimerase
o oligonucleótido ARN/ADN capturado nos cristais adopta uma conformação em forma A, como esperado. O domínio polegar envolve múltiplas interacções com o iniciador de ARN, tanto através de contactos hidrofóbicos como de interacções polares (Perera et al., 2013)., Experimentos em solução têm mostrado que a extensão do iniciador de RNA por pol α é limitada a 10-12 nucleótidos, o que equivale a uma volta de uma hélice. Esta observação levou os autores a sugerir um mecanismo para a resolução do primer síntese por pol α em que a perda de interações específicas entre o polegar e o RNA seria acionar o polimerase para desengatar a partir do DNA/RNA oligonucleotide, e permitir uma mão para uma replicação polimerase.,
movimentos no domínio da Palma podem facilitar a translocação de Pol α
tendo cristalizado a enzima em três estados (apo, binário e ternário) permitiu que os autores sobrepusessem todos os três modelos estruturais. Pol α é a única polimerase de DNA da família eucariótica B para a qual todos os três estados foram capturados em uma estrutura cristalina. A superposição estrutural revelou que, além dos movimentos bem documentados dos dedos e subdomínios do polegar que acompanham a ligação do substrato e a transferência nucleotidila, a própria subdominação da palma sofre um rearranjo estrutural (Perera et al.,, 2013). Os autores propõem que as diferentes conformações do domínio da palma poderiam facilitar a translocação de pol α Ao longo e além do ARN/DNA duplex. Como mencionado acima, a perda de contatos com a cadeia de RNA é prevista para desencadear a liberação de primer, que então se torna disponível para extensão por pol δ Ou ε.,
Uma Proteína Diferentes Dobra no Inativada Exonuclease Subdomínio
A atividade de revisão é abolida em pol α, devido a mutações em todos os quatro carboxylates (Asp114/Glu116/Asp222/Asp327 em RB69 gp43 correspondem a Ser542/Gln544/Tyr644/Asn757 em uma estrutura baseada em alinhamento) (Tabela 2). Além disso, o β-hairpin tema encontrado na maioria das polimerases da família B (resíduos 246-267 em RB69 gp43) é substituído por um helicoidais região pol α (resíduos 667-676; 681-693) (Hogg et al., 2007)., O alfinete β faz parte do domínio das exonucleases e foi demonstrado em T4 e RB69 pols a participação no particionamento do primário do DNA entre a polimerase e o local ativo da exonuclease (Reha-Krantz, 1988; Stocki et al., 1995; Hogg et al., 2007). Na ausência de actividade de revisão, não é surpreendente que este motivo não tenha sido mantido em pol α. Resíduos de sua 684 e Phe 685 da região helicoidal em Pol α stack com uma base de timina e guanina, respectivamente, nas posições-3 e -2 na 5’end não emparelhada do modelo (Perera et al., 2013)., Assim, em pol α, a região correspondente ao motivo do alfinete-β adopta uma dobra diferente (hélices vs. cordões β) e uma função diferente (estabilizando a região não emparelhada do fio-Modelo em vez de facilitar a comutação activa do local). Uma vez que a pol α é desprovida de actividade de revisão, coloca-se a questão de saber se os oligonucleótidos curtos são corrigidos e, em caso afirmativo, por que polimerase de ADN. Parece que a revisão dos primers sintetizados por pol α é realizada por pol δ (Pavlov et al., 2006a).,
a polimerase ADN δ
a pol humana δ é composta por quatro subunidades, enquanto a Saccharomyces cerevisiae tem três (Gerik et al., 1998; Liu et al., 2000) (Quadro 1). Para além da sua função na replicação do ADN, foi demonstrado que o pol δ desempenha um papel na Reparação e recombinação do ADN (Hubscher et al., 2002; Lee et al., 2012; Tahirov, 2012). P12, a menor subunidade no pol humano δ e também a subunidade que não é vista na levedura em crescimento, é degradada em resposta a danos no DNA (Lee et al., 2014). A subunidade catalítica da levedura pol δ (POL3) é composta por 1097 resíduos., A construção utilizada para cristalização inclui resíduos 67-985 e, portanto, não tem o CTD (Figura 1; Quadro 2).
A Third Metal Ion in the Polymerase Active Site
The palm domain contains three conserved carboxylates (Asp608, Asp762, and Asp764). Os dois aspartatos catalíticos, Asp608 e Asp764, entram em contato com dois íons metálicos (Ca2+) no local ativo da polimerase, separados por 3,7 Å. Curiosamente, um terceiro metal foi observado coordenado pelo fosfato γ Do nucleótido de entrada e Glu802, com Glu800 nas proximidades., A mutação de ambos os glutamatos para a alanina produziu uma variante da polimerase com uma eficiência de incorporação reduzida tanto para nucleótidos correctos como incorrectos (Swan et al., 2009). Estes aminoácidos posições, pol α e pol ε também tem carboxilato de etilo resíduos (pol δ Glu800/Glu802 correspondem a pol α Asp1033/Asp1035, e pol ε Glu945/Asp947). Deve investigar-se se estes carboxilatos desempenham papéis semelhantes em pol α e ε.,
high Fidelity and Proofreading
Human pol δ is a high-fidelity polymerase, catalyzing the nucleotidyl transfer reaction with an error frequency of 1 per 22,000 (Schmitt et al., 2009). A revisão aumenta a fidelidade da polimerase por um fator de 10-100 (Mcculloch e Kunkel, 2008; Prindle et al., 2013). Pol δ possui um local ativo de polimerase e exonuclease, separado por cerca de 45 Å (Swan et al., 2009). As ADN polimerases com actividade de revisão são capazes de detectar nucleótidos mal incorporados, contactando o sulco menor de pares de bases para além do local de inserção., A proteína interage com aceitadores universais de ligação de hidrogênio nas posições N3 e O2 de purinas e pirimidinas, respectivamente (Seeman et al., 1976; Doublié et al., 1998; Franklin et al., 2001). Estes contatos de ligação de hidrogênio são preservados quando o par base adota uma geometria Watson-Crick e se perde no caso de um desfasamento. Na gp43 RB69, os contatos se estendem para os dois primeiros pares de base além do recém-nascido par de base (Franklin et al., 2001; Hogg et al., 2004, 2005). Os contactos são muito mais extensos em pol δ, estendendo-se a cinco pares de bases Pós-inserção (Swan et al.,, 2009), que poderia contribuir para a sua alta fidelidade.
Como mencionado acima, o segmento β-hairpin do domínio da exonuclease desempenha um papel crítico na partição do DNA entre locais de polimerização e de revisão em T4 e RB69 pols (Stocki et al., 1995; Hogg et al., 2007). Na RB69 gp43 o tema β-hairpin adota diferentes conformações, dependendo se o complexo foi obtido com DNA não danificado (Franklin et al., 2001; Zahn et al., 2007) ou DNA contendo um dano (Freisinger et al., 2004; Hogg et al., 2004)., Ele foi totalmente visualizado entrando em contato com os fios de primer e template em um complexo com thymine glicol (Aller et al., 2011). Da mesma forma, o β gancho pol δ projeta-se para o major groove do DNA e atua como uma cunha entre o double-stranded DNA e o single-stranded 5’end do modelo vertente, que é estabilizado por dois resíduos aromáticos Phe441 e Tyr446 (Figura 1) (Swan et al., 2009). A posição do gancho β é consistente com um papel na mudança de local ativo.,
Contactos Interdomínicos e fidelidade
mutações envolvidas no cancro são principalmente encontrados no domínio da exonuclease de pol δ e ε, enfatizando o papel crítico da revisão na redução da incidência de mutações (Church et al., 2013; Henninger and Pursell, 2014). Uma mutação nas células cancerígenas colorectais humanas localiza-se no domínio dos dedos, R689W. a mutação análoga na levedura (R696W) resulta num fenótipo mutador (Daee et al., 2010)., Uma mutação na vizinhança de Arg696 no motivo altamente conservado do subdomínio dos dedos da levedura pol δ (a699q) também resulta num fenótipo mutador. Esta região dos dedos está muito próxima da DNT. Mutação Met540 da NTD da alanina, suprime a mutator fenótipo de A699Q, ilustrando que as interações entre os dedos e a NTD pode afetar a fidelidade da polimerase (Prindle et al., 2013)., Similarmente em T4 e RB69 pols, o motivo principal da NPL, que envolve resíduos dos domínios N-terminal e palm, está em contato com o domínio dos dedos e foi mostrado para estabilizar complexos de DNA polimerase (Li et al., 2010).
DNA Polimerase ε
A subunidade catalítica da DNA polimerase ε é o produto de uma grande gene (2222 aminoácidos na levedura; 2286 em humanos), e é apenas o terceiro em tamanho depois de polimerase ζ (que também é um membro da família B) e pol θ, uma família, Uma polimerase (3130 e 2590 aminoácidos, respectivamente, em seres humanos) (Lange et al.,, 2011; Hogg e Johansson, 2012) (Figura 1; Quadro 2). Pol ε É duas vezes maior que pol δ e é composto por duas regiões de polimerase/exonuclease tandem. O segmento N-terminal abriga actividades de polimerase e de revisão, enquanto o segmento C-terminal está inactivado. Os dois módulos de polimerase-exonuclease estão distantemente relacionados (Tahirov et al., 2009). Embora se presume que o segmento inactivado desempenhe um papel estrutural durante a replicação, dois grupos foram capazes de cristalizar construções catalisaticamente ativas de pol ε (resíduos 1-1228; 1-1187) sem todo o módulo C-terminal (Hogg et al.,, 2014; Jain et al., 2014a). Ambas as estruturas cristalinas eram de um complexo ternário da polimerase, DNA primer / template e nucleotídeo de entrada.
Um novo domínio de Processividade que emana do domínio da palma
pol ε difere de pol δ Na medida em que não requer o ADN pinça PCNA para alta processividade (Hogg e Johansson, 2012). O domínio de Palma de Pol ε é substancialmente maior (380 resíduos) do que o de pol α Ou δ (175 e 203 resíduos, respectivamente)., As estruturas cristalinas recentes de pol ε revelaram que as inserções no domínio da palma formam colectivamente um novo domínio composto por três cadeias β e duas hélices (resíduos 533-555; 682-760) (Hogg et al., 2014; Jain et al., 2014a) (Figura 1; Quadro S1). A remoção de resíduos 690-751 resultou numa variante com diminuição da actividade da polimerase. Além disso, a mutação de resíduos positivamente carregados (His748, Arg749 e Lys751) localizados nas proximidades da espinha dorsal de fosfato afetou a processividade da enzima (Hogg et al., 2014)., O domínio extra originário da palma foi assim chamado de processividade ou domínio P, após sua função. A base do domínio P abriga um local de ligação de metal (ver abaixo) (Hogg et al., 2014; Jain et al., 2014a, b).
um aglomerado de enxofre de ferro dentro do domínio da polimerase
inesperadamente estudos de solução revelaram que a subunidade catalítica da polimerase ε contém um aglomerado dentro da sua dobra de polimerase (Jain et al., 2014b), para além do agregado no CTD (Figura 2; Quadro 2)., O segundo aglomerado dentro de Pol ε sugere que esta polimerase pode ser mais sensível ao estresse oxidativo (Jain et al., 2014b). As estruturas cristalinas de Pol ε, No entanto, não revelaram um aglomerado no domínio da polimerase (Hogg et al., 2014; Jain et al., 2014a; Zahn and Doublié, 2014). Dois dos resíduos de cisteínas estão desordenados nos modelos estruturais e o local de ligação do metal resultante parece ligar o zinco (Hogg et al., 2014; Jain et al., 2014a). A substituição de A por um zinco não nativo em proteínas de ligação metal não é incomum (Netz et al., 2012) as clusters are labile., A visualização no domínio da polimerase de pol ε pode exigir condições anaeróbias.
A Short β-Hairpin Motif in the Exonuclease Domain
In any DNA polymerase harboring both polymerase and exonuclease activities the bound DNA is in equilibrium between the two active centers (Beechem et al., 1998). A concentração de nucleótido recebido e a presença de uma base danificada ou emparelhada são dois fatores que influenciam a transferência de DNA do local de ativação da polimerase para o local ativo de revisão., As polimerases monitoram o lado sulco menor dos recém-formados pares de base e interagem com os aceitadores universais h bond, O3, e N2, como uma forma de verificar se há discrepâncias (Seeman et al., 1976; Franklin et al., 2001). Uma característica única de Pol ε é o contato com o maior lado sulco do par base nascente através de um resíduo do domínio da exonuclease, Tyr431. É necessária uma análise mais aprofundada para elucidar o papel potencial desta tirosina na alta fidelidade de pol ε.,
In pol δ The β-hairpin segment inserts itself in the DNA and acts as a wedge between single-stranded and double-stranded DNA (Swan et al., 2009). In E. coli DNA pol II, the insertion of a β barrel shops the position of The β hairpin in such a way that polymerization is favoured over proofreading (Wang and Yang, 2009). Esta modificação, presumivelmente, permite que esta polimerase realize a extensão da síntese de translesões., Desde pol ε é preciso DNA polimerase a suposição de que a estrutura de cristal seria a de que o β gancho de cabelo deve ser mais semelhante ao de pol δ do que o da E. coli Pol II. Surpreendentemente, o β-hairpin motivo pol ε é truncado, muito curto para contato com o DNA (Figura 1). Que motivo proteico, então, pode estar a facilitar a mudança activa do local ao sentir um erro? O domínio P é um bom candidato, por causa de seus contatos com as cadeias de primer e template; resíduos do domínio P poderiam sentir erros de replicação e, assim, podem ajudar a facilitar a comutação do site ativo.,
Conclusões
os três eucarióticas replicação do DNA polimerases uso comum B-família dobra, e cada polimerase incorporou modificado elementos estruturais que são exclusivos e sob medida para cada polimerase da função específica (por exemplo, a adição de processivity domínio pol ε, um processive polimerase, que não use o PCNA, ou a modificação região de contato com o 5’end do modelo pol α, uma polimerase desprovido de revisão de actividade). A dobra de polimerases da família B é adequada para polimerases de alta fidelidade e replicação., Mas surpreendentemente, também é usado por polimerases de translação. Eucariótico pol ζ (Ou REV3L) é uma polimerase 353 kDa que funciona em síntese de translação e parece suprimir a tumorigénese (Wittschieben et al., 2010; Lange et al., 2011; Zahn et al., 2011; Hogg and Johansson, 2012; Sharma et al., 2013). A estrutura da E. coli Pol II revelou modificações na DNT que afetam a posição da cavilha β do domínio da exonuclease, e assim particionamento do DNA entre os locais de polimerização e revisão (Wang e Yang, 2009)., A estrutura de Pol ζ pode revelar ajustes similares, que alteram a dobra utilizada por polimerases replicativas de alta fidelidade para tornar a enzima menos fiel e capaz de realizar a síntese de translação.
Declaração de conflito de interesses
os autores declaram que a investigação foi realizada na ausência de quaisquer relações comerciais ou financeiras que possam ser interpretadas como um potencial conflito de interesses.
reconhecimento
Este trabalho foi apoiado por uma subvenção dos Institutos Nacionais de saúde (NCI R01 CA 52040).material suplementar
















Deixe uma resposta