The completion of The Human Genome Project in 2003 ushered in a new era of rapid, affordable, and accurate genome analysis—called Next Generation Sequencing (NGS). A NGS baseia-se em tecnologias de” sequenciamento de primeira geração ” para produzir resultados de sequenciamento precisos e econômicos.Fred Sanger sequenciou o primeiro genoma completo do ADN, o vírus phage ?X174, em 1977., Nesse mesmo ano, Sanger desenvolveu a futura espinha dorsal da era do genoma: tecnologia de sequenciamento de DNA. Sua técnica usou o método de terminação de cadeia. Isso agora é visto como a “tecnologia de primeira geração” da sequenciação do genoma.
Uma vez que a sequenciação de Sanger (ou o método de terminação de cadeia) é a primeira geração de tecnologia de sequenciamento, entendendo que é muito importante. O Projeto Genoma Humano começou com a tecnologia de sequenciamento de Sanger.,
The Sanger Sequencing Method
The Sanger sequencing method reles on dideoxynucleotides(ddNTPs), a type of deoxynucleoside triphosphates (dNTPs), that lack a 3′ hydroxyl group and have a hydrogen atom instead . Quando essas bases se ligam à sequência de DNA crescente, elas terminam a replicação como elas não podem se ligar a outras bases. Para executar sequenciamento de Sanger, você adiciona seus iniciadores a uma solução contendo a informação genética a ser sequenciada, em seguida, dividir a solução em quatro reações PCR., Cada reação contém uma mistura dNTP com um dos quatro nucleótidos substituídos por um ddntp (a, T, G, E C ddntp grupos). No final da PCR, cada uma das suas quatro reações produzirá produtos PCR de vários comprimentos porque a replicação é interrompida aleatoriamente. Ao executar as amostras em um gel com 4 faixas, você pode juntar a sequência como cada sequência foi replicada a partir do mesmo material original. Aqui está um exemplo onde os ddNTPs estão em negrito e os dNTPs não são:
sua sequência é ATGCTCAG.,
Os quatro reações dar-lhe:
Reação com ddATP: UM, ATGCTCA, ATGCTCAG
Reação com ddGTP: ATG, ATGCTCAG
Reação com ddCTP: ATGC, ATGCTC, ATGCTCAG
Reação com ddTTP: NÃO, ATGCT, ATGCTCAG
Todas as reações depois de executar um gel seria algo parecido com isto (Imagem por Olwen Reina):
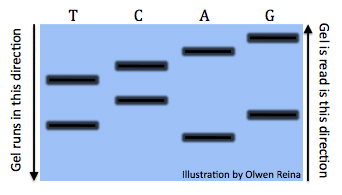
Cada banda denota os diferentes comprimentos de código. Por exemplo, a banda é a direita sob o “A” simboliza a sequência: “ATGCTCA”
ainda confusa?vamos imaginar um jogo de festa. O jogo é um jogo de adivinhas., Aqui está como ele é jogado:
Você está pensando em um número e o grupo tem que adivinhar. A parte complicada é que o número tem 200 dígitos de comprimento. Você está lendo os dígitos do número em sua cabeça sem fazer um som. De vez em quando uma pessoa interrompe você, e você diz a eles o único dígito que você estava apenas pensando e onde ele está na seqüência de 200. Cada vez que você é interrompido, você tem que começar de novo. Você sai depois de algumas horas e o grupo tem que descobrir o número de 200 dígitos., Eles têm que juntar as informações que você deu a eles, por exemplo, o 25º número ERA 5, o 40º número ERA 0, e assim por diante. Usando a informação de suas interrupções, eles podem repetir o número que lhe deram.
embora este pareça o jogo mais lamechas do mundo, funciona muito bem para sequenciação!
infelizmente, é lento, caro, e (anteriormente) depende de materiais radioativos. Isto levou os cientistas a desenvolver novas e melhores formas de sequenciamento do genoma.,os maiores avanços na sequenciação do genoma têm sido o aumento da velocidade e precisão, resultando na redução de mão-de-obra e custo. A velocidade é graças à análise paralela e Tecnologia de alta produtividade. É a diferença entre ter uma criança de 4 anos a ler Moby Dick e dar um parágrafo Cada um a uma série de estudantes de teatro e pedir-lhes para lerem imediatamente.as máquinas Sequenciadoras melhoraram desde que Sanger desenvolveu o seu método., Em 1987, a Applied Biosystems tornou-se a primeira a introduzir uma máquina de sequenciação automática, chamada AB370. A máquina se baseou em um método chamado eletroforese capilar que usou o método de terminação de cadeia de Sanger sem precisar de um gel.na mistura de reacção PCR adicionam-se ddNTPs de terminação em cadeia com fluorescência. Pelo método de sequenciamento de Sanger, produtos PCR de vários comprimentos são criados, e então separados de acordo com seu tamanho. O tamanho é medido pela carga negativa global do produto PCR. Quanto mais negativa a carga, maior o fragmento., Tome o exemplo acima da incorporação de ddNTP e imagine que as letras a negrito são ddNTPs com um fluorocromo ligado. Como os fragmentos são puxados para o eletrodo positivo de um capilar (ver imagem abaixo), eles passam por um feixe laser que desencadeia um flash de luz do fluorocromo ligado ao ddNTP que é característica do tipo base (por exemplo, verde para a, amarelo para T, azul para G, vermelho para C). Desta forma, o genoma é cuidadosamente lido.
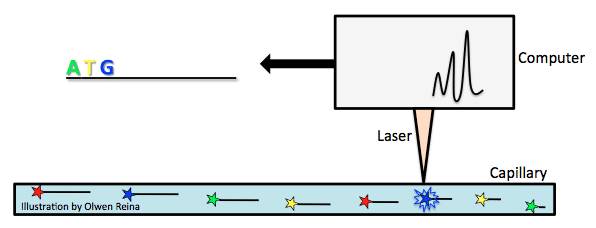
A maior diferença aqui foi a velocidade e o custo., AB370 poderia detectar 96 bases de uma só vez, 500k bases por dia, e tinha um comprimento de leitura de 600 bases usando uma análise paralela e configuração de alta produtividade. Esta forma de sequenciamento tornou-se a principal ferramenta para a conclusão do Projeto Genoma Humano.
Como É que os NGS se comparam com a sequenciação de primeira geração?
NGS é caracterizado por maior precisão e velocidade, mas também redução de mão de obra e custo. Nunca houve uma época em que fosse tão barato, conveniente ou direto sequenciar um genoma., Indiscutivelmente, a maior melhoria tem sido o desenvolvimento de análises paralelas, que aumentaram a velocidade sequenciadora.
a única coisa que nos atrasa agora é a interpretação dos resultados!

isto ajudou-o? Então, por favor, partilhe com a sua rede.
















Deixe uma resposta