Introducción
la replicación en el núcleo de células eucariotas emplea tres polimerasas de ADN: polimerasa α, δ y ε (Hubscher et al., 2002; Pavlov et al., 2006B; Kunkel y Burgers, 2008; Loeb y Monnat, 2008; Burgers, 2009; Pavlov y Shcherbakova, 2010; Lange et al., 2011)., La síntesis de ADN es direccional y procede de 5′ a 3′, donde el ataque nucleofílico sobre el fosfato α de un nucleótido por el 3’OH de un cebador resulta en la incorporación de un nucleósido monofosfato y la liberación de pirofosfato (Steitz, 1999). Todas las polimerasas de ADN requieren una imprimación y un 3’OH libre para conducir la síntesis de ADN, y Pol α No es una excepción. Pol α es un heterotetrámero compuesto por dos subunidades primasas y dos subunidades polimerasas., Las subunidades primasas inician la replicación del ADN sintetizando cebadores de ARN cortos (7-12 ribonucleótidos), que luego son extendidos por la polimerasa α (Pellegrini, 2012). La polimerasa de ADN δ y ε alargan los cebadores generados por pol α de manera precisa y procesiva (Kunkel, 2004, 2011; Pellegrini, 2012). En la levadura, se ha demostrado que la ADN polimerasa δ es esencial para la síntesis de ADN de la hebra rezagada, mientras que pol ε parece funcionar principalmente en la hebra principal (Pursell et al., 2007; Nick Mcelhinny et al., 2008; Kunkel, 2011; Georgescu et al., 2014)., En contraste, en la replicación mitocondrial es responsabilidad de una única polimerasa, la polimerasa γ DEL ADN(Lee et al., 2009).
las polimerasas de ADN se agrupan en siete familias (A, B, C, D, X, Y y RT). En los eucariotas, las tres polimerasas replicativas nucleares del ADN pertenecen a la familia B (Burgers et al., 2001; Patel y Loeb, 2001). Ahora hay estructuras de cristales de las tres polimerasas de ADN replicativas de levadura, que permiten por primera vez una comparación de sus elementos estructurales compartidos, así como un estudio de sus características únicas (Swan et al., 2009; Perera et al.,, 2013; Hogg et al., 2014; Jain et al., 2014a). Las tres ADN polimerasas replicativas son enzimas multi-subunidades (Tabla 1) (Johansson y Macneill, 2010; Pavlov y Shcherbakova, 2010; Makarova et al., 2014). El foco principal de esta revisión está en su dominio catalítico, o subunidad A.
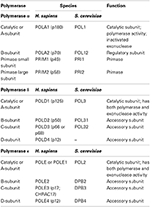
Tabla 1. Las ADN polimerasas eucariotas son enzimas multi-subunidades.,
estructura general de las polimerasas de la familia b
todas las polimerasas de ADN comparten un pliegue de polimerasa común, que se ha comparado con una mano derecha humana, compuesta de tres subdominios: dedos, palma y pulgar (Steitz, 1999; Patel y Loeb, 2001). La palma, un pliegue altamente conservado compuesto por cuatro filamentos β antiparalelos y dos hélices, alberga dos aspartatos catalíticos estrictamente conservados ubicados en el motivo a, DXXLYPS y el motivo C, DTDS (Delarue et al., 1990; Braithwaite e It, 1993)., Este pliegue similar al RRM es compartido por un grupo muy grande de enzimas, incluyendo ADN y ARN polimerasas, transcriptasas inversas, polimerasa CRISPR e incluso transferasas inversas (3′-5′) como Thg1 (Anantharaman et al., 2010; Hyde et al., 2010). En contraste, los subdominios del pulgar y los dedos exhiben una diversidad estructural sustancialmente mayor (Steitz, 1999). Los dedos experimentan un cambio conformacional al unir el ADN y el nucleótido entrante correcto. Este movimiento permite que los residuos en el subdominio de los dedos entren en contacto con el nucleótido en el par de bases nacientes., El pulgar sostiene el duplex de ADN durante la replicación y juega un papel en la processividad (Doublié y Ellenberger, 1998; Doublié et al., 1999).
las polimerasas de ADN eucariotas α, δ y ε comparten homología con muchas polimerasas arqueales, bacterianas, bacteriófagas y virales (Delarue et al., 1990; Braithwaite e It, 1993; Franklin et al., 2001; Firbank et al., 2008; Wang y Yang, 2009)., Koonin y colaboradores contribuyeron con un análisis filogenético detallado de las polimerasas del ADN arqueal y su relación con las polimerasas eucariotas en este número de Frontiers in Microbiology dedicado a las polimerasas Makarova et al. (2014).
todas las polimerasas de la familia B se componen de cinco subdominios: los dedos, el pulgar y la palma (descritos anteriormente) que constituyen el núcleo de la enzima, así como un dominio exonucleasa y un dominio N-terminal (NTD) (Franklin et al., 2001; Xia y Konigsberg, 2014) (Figura 1; Cuadro S1)., El dominio exonucleasa lleva una actividad de corrección de 3′-5′, que elimina los nucleótidos mal incorporados. El sitio activo de la exonucleasa se encuentra a 40-45 Å del sitio activo de la polimerasa. El NTD parece carecer de actividad catalítica. En Pol δ el NTD comprende tres motivos: uno tiene una topología que se asemeja a un pliegue OB, un motivo de unión de ADN de una sola cadena, y otro tiene un motivo de unión de ARN (motivo de reconocimiento de ARN o RRM) (Swan et al., 2009). En el bacteriófago T4, las mutaciones en el NTD disminuyen la expresión de la polimerasa (Hughes et al., 1987)., En RB69 y T4, la polimerasa gp43 se une a su propio ARN mensajero, presumiblemente a través de la NTD y reprime la traducción (Petrov et al., 2002), que no parece ser el caso de pol δ (Swan et al., 2009). Nuevos datos indican que el NTD juega un papel en la estabilidad y fidelidad de la polimerasa a través de sus interacciones con otros dominios (Li et al., 2010; Prindle et al., 2013) (véase INFRA).
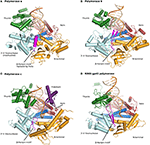
la Figura 1. Los complejos ternarios de polimerasas α, δ, ε y rb69 gp43 se ilustran a partir de orientaciones idénticas para la comparación., Los dominios pulgar (verde) y dedos (azul oscuro) agarran el ácido nucleico dúplex (imprimación mostrada en beige, plantilla en naranja) contra el dominio palma (rojo). El dominio N-terminal aparece en oro, adyacente al dominio exonucleasa 3 ‘ -5 ‘ (cian). (A) la polimerasa α (PDBID 4FYD) se une a un híbrido ARN/ADN, donde el surco menor ancho y poco profundo del ADN en Forma A es aparente cerca del pulgar. El dominio exonucleasa 3′-5’ está desprovisto de actividad. Una región helicoidal (magenta) en el dominio exonucleasa inactivada estabiliza el 5’end de la plantilla., B) la polimerasa δ (PDBID 3iay) alberga un gran motivo de horquilla β (magenta), que es importante para cambiar la hebra de imprimación del sitio activo de la polimerasa al sitio activo de la exonucleasa en caso de revisión. (C) la polimerasa ε (PDBID 4M8O) ejerce un dominio P único (púrpura), que dota a la polimerasa de mayor processividad. Curiosamente, el motivo β horquilla está atrofiado en Pol ε., D) la conservación del pliegue de la polimerasa del ADN de la familia B, y la organización del dominio, es evidente cuando se observa la enzima modelo del bacteriófago rb69 gp43 (PDBID 2OZS) junto con las tres polimerasas replicativas eucariotas. La delineación del dominio para cada polimerasa se da en la tabla S1. La figura fue hecha con PyMOL (el sistema de gráficos moleculares PyMOL, versión 1.5.0.4 Schrödinger, LLC.).
se sabe que todas las ADN polimerasas de la familia B de mamíferos albergan dos sitios de unión de metales ricos en cisteína (CysA y CysB) en su dominio C-terminal (CTD) (Figura 2)., Se presume que CysA es un sitio de unión al zinc, mientras que CysB es un cúmulo de hierro y azufre(Netz et al., 2012). La pérdida del clúster en la CTD de la levadura Pol δ afecta negativamente las interacciones con su subunidad B accesoria (Sánchez García et al., 2004). Se demostró que el motivo de unión de zinc es importante para la interacción de Pol δ con su factor de processividad, PCNA (Netz et al., 2012).

la Figura 2. Diagrama esquemático de las tres polimerasas de ADN replicativas α, δ y ε de Saccharomyces cerevisiae., La ADN polimerasa del bacteriófago rb69 se muestra para la comparación.
ADN Polimerasa α
La subunidad catalítica de la ADN polimerasa α se compone de 1468 aminoácidos (Tabla 2). El constructo proteico diseñado para la cristalización fue truncado en los terminales n y C (residuos 349-1258) y por lo tanto carece de la CTD y su cúmulo (Figura 2). El constructo fue cristalizado en un complejo binario con un oligonucleótido híbrido ADN/ARN, y en un complejo ternario con ADN / ARN y nucleótido entrante (Perera et al., 2013) (gráfico 1).,

la Tabla 2. Compilation of DNA polymerases of the B family of known structure.
un mecanismo de desconexión de la polimerasa
el oligonucleótido de ARN/ADN capturado en los cristales adopta una conformación en forma A, como se esperaba. El dominio pulgar participa en múltiples interacciones con el primer de ARN, tanto a través de contactos hidrofóbicos como de interacciones polares (Perera et al., 2013)., Los experimentos en solución han demostrado que la extensión de la imprimación de ARN por pol α se limita a 10-12 nucleótidos, lo que equivale a una vuelta de una hélice. Esta observación llevó a los autores a sugerir un mecanismo para la terminación de la síntesis de imprimación por Pol α en el que la pérdida de interacciones específicas entre el pulgar y el ARN desencadenaría la polimerasa para desacoplarse del oligonucleótido ADN/ARN, y permitir una mano a una polimerasa replicativa.,
los movimientos en el dominio Palm pueden facilitar la translocación de Pol α
haber cristalizado la enzima en tres estados (apo, binario y ternario) permitió a los autores superponer los tres modelos estructurales. Pol α es la única polimerasa de ADN de la familia B eucariota para la cual los tres estados fueron capturados en una estructura cristalina. La superposición estructural reveló que, además de los movimientos bien documentados de los subdominios de los dedos y el pulgar que acompañan la Unión del sustrato y la transferencia de nucleotidilo, el subdominio de la palma en sí sufre un reordenamiento estructural (Perera et al.,, 2013). Los autores proponen que las diferentes conformaciones del dominio palm podrían facilitar la translocación de pol α a lo largo y más allá del dúplex ARN/ADN. Como se mencionó anteriormente, se predice que la pérdida de contactos a la cadena de ARN desencadenará la liberación de imprimación, que luego estará disponible para su extensión por pol δ O ε.,
un pliegue proteico diferente en el subdominio de exonucleasa inactivada
la actividad de revisión se suprime en pol α, debido a mutaciones en los cuatro carboxilatos (Asp114/Glu116/Asp222/Asp327 en rb69 gp43 corresponden a Ser542/Gln544/Tyr644 / Asn757 en una alineación basada en la estructura) (Tabla 2). Además, el motivo β-horquilla encontrado en la mayoría de las polimerasas de la familia B (residuos 246-267 en rb69 gp43) es reemplazado por una región helicoidal en Pol α (residuos 667-676; 681-693) (Hogg et al., 2007)., La horquilla β es parte del dominio exonucleasa y se ha demostrado en las pols T4 y RB69 que participa en la partición de la cartilla de ADN entre la polimerasa y el sitio activo de exonucleasa (Reha-Krantz, 1988; Stocki et al., 1995; Hogg et al., 2007). En ausencia de la actividad de corrección no es sorprendente que este motivo no se mantuvo en Pol α. Residuos His 684 y Phe 685 de la región helicoidal en la pila Pol α con una base de timina y guanina, respectivamente, en las posiciones -3 y -2 en el 5 ‘ final no emparejado de la plantilla (Perera et al., 2013)., Por lo tanto, en Pol α la región correspondiente al motivo β-horquilla adopta un pliegue diferente (hélices vs.hebras β) y una función diferente (estabilizar la región no emparejada de la hebra de la plantilla en lugar de facilitar el cambio de sitio activo). Dado que pol α carece de actividad de revisión, surge la pregunta de si los oligonucleótidos cortos se corrigen y, en caso afirmativo, por qué ADN polimerasa. Parece que la corrección de los cebadores sintetizados por pol α es realizada por Pol δ (Pavlov et al., 2006a).,
ADN polimerasa δ
La Pol δ humana está compuesta de cuatro subunidades, mientras que Saccharomyces cerevisiae tiene tres (Gerik et al., 1998; Liu et al., 2000) (Cuadro 1). Además de su función en la replicación del ADN, se ha demostrado que pol δ desempeña un papel en la reparación y recombinación del ADN (Hubscher et al., 2002; Lee et al., 2012; Tahirov, 2012). P12, la subunidad más pequeña en Pol δ humano y también la subunidad que no se ve en la levadura en ciernes, se degrada en respuesta al daño del ADN (Lee et al., 2014). La subunidad catalítica de la levadura Pol δ (POL3) se compone de 1097 residuos., El constructo utilizado para la cristalización comprende residuos 67-985 y por lo tanto carece del CTD (Figura 1; Tabla 2).
un tercer Ion metálico en el sitio activo de la polimerasa
el dominio palm contiene tres carboxilatos conservados (Asp608, Asp762 y Asp764). Los dos aspartatos catalíticos, Asp608 y Asp764, entran en contacto con dos iones metálicos (Ca2+) en el sitio activo de la polimerasa separados por 3,7 Å. Curiosamente se observó un tercer metal coordinado por el fosfato γ del nucleótido entrante y Glu802, con Glu800 en las proximidades., La mutación de ambos glutamatos a alanina produjo una variante de polimerasa con menor eficiencia de incorporación tanto para nucleótidos correctos como incorrectos (Swan et al., 2009). En estas posiciones de aminoácidos, pol α y Pol ε también tienen residuos de carboxilato (Pol δ Glu800/Glu802 corresponden a Pol α Asp1033/Asp1035, y pol ε Glu945/Asp947). Queda por investigar si estos carboxilatos desempeñan papeles similares en Pol α y ε.,
alta fidelidad y corrección
La Pol δ humana es una polimerasa de alta fidelidad, catalizando la reacción de transferencia de nucleótidos con una frecuencia de error de 1 por 22.000 (Schmitt et al., 2009). La revisión aumenta la fidelidad de la polimerasa en un factor de 10-100 (Mcculloch y Kunkel, 2008; Prindle et al., 2013). Pol δ alberga un sitio activo de polimerasa y exonucleasa, separados por aproximadamente 45 Å(Swan et al., 2009). Las polimerasas de ADN con actividad de revisión son capaces de detectar nucleótidos mal incorporados al entrar en contacto con el surco menor de pares de bases más allá del sitio de inserción., La proteína interactúa con los aceptadores universales de enlaces de hidrógeno en las posiciones N3 y o2 de purinas y pirimidinas, respectivamente (Seeman et al., 1976; Doublié et al., 1998; Franklin et al., 2001). Estos contactos de enlace de hidrógeno se conservan cuando el par de bases adopta una geometría Watson-Crick y se pierden en caso de un desajuste. En rb69 gp43, los contactos se extienden a los dos primeros pares de bases más allá del par de bases naciente (Franklin et al., 2001; Hogg et al., 2004, 2005). Los contactos son mucho más extensos en Pol δ, extendiéndose a cinco pares de bases después de la inserción(Swan et al.,, 2009), que podría contribuir a su alta fidelidad.
como se mencionó anteriormente, el segmento β-horquilla del dominio exonucleasa juega un papel crítico en la partición del ADN entre los sitios de polimerización y revisión en las pols T4 y RB69 (Stocki et al., 1995; Hogg et al., 2007). En rb69 gp43 el motivo β-horquilla adopta diferentes conformaciones, dependiendo de si el complejo se obtuvo con ADN no dañado (Franklin et al., 2001; Zahn et al., 2007) o el ADN que contiene un daño (Freisinger et al., 2004; Hogg et al., 2004)., Se visualizó completamente el contacto de las hebras de imprimación y plantilla en un complejo con timina glicol (Aller et al., 2011). De manera similar, la horquilla β en Pol δ sobresale en el surco principal del ADN y actúa como una cuña entre el ADN de doble cadena y el 5’end de una sola cadena de la hebra plantilla, que se estabiliza por dos residuos aromáticos Phe441 y Tyr446 (Figura 1) (Swan et al., 2009). La posición de la horquilla β Es consistente con un papel en el cambio de sitio activo.,
contactos Interdomanos y fidelidad
Las mutaciones involucradas en el cáncer se encuentran principalmente en el dominio exonucleasa de Pol δ y ε, enfatizando el papel crítico de la revisión en la reducción de la incidencia de mutaciones (Church et al., 2013; Henninger y Pursell, 2014). Una mutación en las células cancerosas colorrectales humanas se localiza en el dominio de los dedos, R689W. la mutación análoga en levadura (R696W) resulta en un fenotipo mutante (Daee et al., 2010)., Una mutación en la vecindad de Arg696 en el motivo B altamente conservado del subdominio de los dedos de la levadura Pol δ (a699q) también resulta en un fenotipo mutante. Esta región de los dedos está muy cerca del NTD. La mutación de Met540 de la DTN a alanina suprime el fenotipo mutante de A699Q, ilustrando que las interacciones entre los dedos y la DTN pueden afectar la fidelidad de la polimerasa (Prindle et al., 2013)., De manera similar, en los pols T4 y rb69, el motivo del núcleo de la NPL, que involucra residuos de los dominios N-terminal y palm, está en contacto con el dominio de los dedos y se demostró que estabiliza los complejos polimerasa-ADN (Li et al., 2010).
ADN polimerasa ε
la subunidad catalítica de la ADN polimerasa ε es el producto de un gen muy grande (2222 aminoácidos en levadura; 2286 en humanos), y es solo la tercera en tamaño después de la polimerasa ζ (también un miembro de la familia B) y pol θ, una polimerasa de la familia a (3130 y 2590 aminoácidos, respectivamente, en humanos) (Lange et al.,, 2011; Hogg y Johansson, 2012) (Figura 1; Tabla 2). Pol ε es dos veces más grande que POL δ y se compone de dos regiones de polimerasa/exonucleasa en tándem. El segmento N-terminal alberga actividades de polimerasa y corrección, mientras que el segmento C-terminal está inactivado. Los dos módulos exonucleasa-polimerasa están distanciadamente relacionados (Tahirov et al., 2009). Aunque se presume que el segmento inactivado desempeña un papel estructural durante la replicación, dos grupos fueron capaces de cristalizar constructos de Pol ε catalíticamente activos (residuos 1-1228; 1-1187) que carecen de todo el módulo C-terminal (Hogg et al.,, 2014; Jain et al., 2014a). Ambas estructuras cristalinas eran de un complejo ternario de la polimerasa, base/plantilla de ADN y nucleótido entrante.
un nuevo dominio de Processividad que emana del dominio Palm
Pol ε difiere de Pol δ en que no requiere la abrazadera deslizante de ADN PCNA para una alta processividad (Hogg y Johansson, 2012). El dominio Palmeral de pol ε es sustancialmente mayor (380 residuos) que el de Pol α O δ (175 y 203 residuos, respectivamente)., Las recientes estructuras cristalinas de Pol ε revelaron que las inserciones en el dominio de la palma forman colectivamente un nuevo dominio que consiste en tres hebras β y dos hélices (residuos 533-555; 682-760) (Hogg et al., 2014; Jain et al., 2014a) (Figura 1; Cuadro S1). La eliminación de residuos 690-751 resultó en una variante con disminución de la actividad de la polimerasa. Además, la mutación de residuos cargados positivamente (His748, Arg749 y Lys751) ubicados en las proximidades de la columna vertebral de fosfato afectó la processividad de la enzima (Hogg et al., 2014)., El dominio extra originario de la palm fue así llamado el dominio processivity O P, Después de su función. La base del dominio P alberga un sitio de unión de metal (ver más abajo) (Hogg et al., 2014; Jain et al., 2014a, b).
un racimo del azufre del hierro dentro del dominio de la polimerasa
inesperadamente los estudios de la solución revelaron que la subunidad catalítica de la levadura polimerasa ε sí mismo contiene un racimo dentro de su doblez de la polimerasa (Jain et al., 2014b), además del clúster en el CTD (Figura 2; Tabla 2)., El segundo grupo dentro de pol ε sugiere que esta polimerasa puede ser más sensible al estrés oxidativo (Jain et al., 2014b). Las estructuras cristalinas de pol ε, Sin embargo, no revelaron un cúmulo en el dominio de la polimerasa (Hogg et al., 2014; Jain et al., 2014a; Zahn y Doublié, 2014). Dos de los residuos de cisteínas están desordenados en los modelos estructurales y el sitio de unión del metal resultante parece unirse al zinc (Hogg et al., 2014; Jain et al., 2014a). La sustitución de A por un zinc no nativo en proteínas de unión a metales no es inusual (Netz et al., 2012) ya que los clusters son lábiles., La visualización dentro del dominio de la polimerasa de Pol ε puede requerir condiciones anaeróbicas.
un motivo corto de β-horquilla en el dominio exonucleasa
en cualquier polimerasa de ADN que alberga actividades tanto de polimerasa como de exonucleasa, el ADN Unido está en equilibrio entre los dos centros activos (Beechem et al., 1998). La concentración de nucleótido entrante y la presencia de una base dañada o de un error de par son dos factores que influyen en la transferencia de ADN desde el sitio de activación de la polimerasa al sitio activo de corrección., Las polimerasas monitorean el lado menor del surco de los pares de bases recién formados e interactúan con los aceptadores de enlace H universales, O3 y N2, como una forma de verificar los desajustes (Seeman et al., 1976; Franklin et al., 2001). Una característica única de pol ε es el contacto con el lado de ranura principal del par de bases naciente a través de un residuo del dominio exonucleasa, Tyr431. Se justifica un análisis adicional para dilucidar el papel potencial de esta tirosina en la alta fidelidad de pol ε.,
en Pol δ el segmento β-horquilla se inserta en el ADN y actúa como una cuña entre el ADN de cadena simple y doble (Swan et al., 2009). En E. coli DNA pol II, la inserción de un barril β cambia la posición de la horquilla β De tal manera que se favorece la polimerización sobre la revisión (Wang y Yang, 2009). Esta modificación permite presumiblemente que esta polimerasa lleve a cabo la extensión de la síntesis de translesión., Dado que pol ε es una polimerasa de ADN precisa, la suposición antes del conocimiento de la estructura cristalina sería que la horquilla β debería estar más cerca de la de Pol δ que la de E. coli Pol II. sorprendentemente, el motivo β-horquilla en pol ε está truncado, demasiado corto para entrar en contacto con el ADN (Figura 1). ¿Qué motivo proteico, entonces, podría facilitar el cambio de sitio activo al detectar un error? El dominio P es un buen candidato, debido a sus contactos con las cadenas de imprimación y plantilla; los residuos del dominio P podrían detectar errores de replicación y, por lo tanto, pueden ayudar a facilitar el cambio de sitio activo.,
conclusiones
Las tres polimerasas replicativas de ADN eucariotas utilizan un pliegue común de la familia B, y cada polimerasa ha incorporado elementos estructurales modificados que son únicos y adaptados para la función específica de cada polimerasa (por ejemplo, la adición del dominio de processividad en pol ε, una polimerasa procesiva que no usa PCNA, o la región modificada que entra en contacto con el 5’end de la plantilla en pol α, una polimerasa desprovista de actividad de revisión). El pliegue de las polimerasas de la familia B es muy adecuado para polimerasas replicativas de alta fidelidad., Pero sorprendentemente, también es utilizado por las polimerasas de translesion. Eukaryotic pol ζ (O REV3L) es una polimerasa de 353 kDa que funciona en la síntesis de translesión y parece suprimir la tumorigenesis (wittschieben et al., 2010; Lange et al., 2011; Zahn et al., 2011; Hogg y Johansson, 2012; Sharma et al., 2013). La estructura de E. coli Pol II reveló modificaciones en el NTD que afectan la posición de la horquilla β del dominio exonucleasa, y por lo tanto la partición del ADN entre los sitios de polimerización y revisión (Wang y Yang, 2009)., La estructura de pol ζ puede revelar ajustes similares, que alteran el pliegue empleado por las polimerasas replicativas de alta fidelidad para hacer que la enzima sea menos fiel y capaz de realizar la síntesis de translesión.
Declaración de conflicto de intereses
los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran ser interpretadas como un potencial conflicto de intereses.
reconocimiento
Este trabajo fue apoyado por una subvención de los Institutos Nacionales de la salud (NCI R01 CA 52040).
















Deja una respuesta