la finalización del proyecto del Genoma Humano en 2003 marcó el comienzo de una nueva era de análisis del genoma rápido, asequible y preciso, llamado Next Generation Sequencing (NGS). NGS se basa en tecnologías de «secuenciación de primera generación» para obtener resultados de secuenciación precisos y rentables.
Fred Sanger secuenció el primer genoma de ADN, los virus fago ?X174, en 1977., En ese mismo año, Sanger desarrolló la futura columna vertebral de la era del genoma: la tecnología de secuenciación de ADN. Su técnica utilizó el método de terminación de la cadena. Esto ahora se ve como la» tecnología de primera generación » de secuenciación del genoma.
dado que la secuenciación de Sanger (o el método de terminación de cadena) es la primera generación de tecnología de secuenciación, entenderla es muy importante. El proyecto del genoma humano comenzó con la tecnología de secuenciación de Sanger.,
El Método de secuenciación de Sanger
El Método de secuenciación de Sanger se basa en dideoxinucleótidos (ddNTPs),un tipo de trifosfatos de desoxinucleósido (dNTPs), que carecen de un grupo hidroxilo 3′ y tienen un átomo de hidrógeno en su lugar . Cuando estas bases se unen a la secuencia de ADN en crecimiento, terminan la replicación ya que no pueden unirse a otras bases. Para realizar la secuenciación de Sanger, agrega sus cebadores a una solución que contiene la información genética a secuenciar, luego divide la solución en cuatro reacciones de PCR., Cada reacción contiene una mezcla con dNTP con uno de los cuatro nucleótidos sustituidos por un ddNTP (grupos ddNTP A, T, G y C). Al final de la PCR, cada una de sus cuatro reacciones producirá productos de PCR de varias longitudes porque la replicación se termina aleatoriamente. Al ejecutar las muestras en un gel con 4 carriles, puede juntar la secuencia ya que cada secuencia se ha replicado del mismo material original. Aquí hay un ejemplo donde los ddNTPs están en negrita y los dNTPs no:
Su secuencia es ATGCTCAG.,
sus cuatro reacciones le dan:
reacción con ddATP: a, ATGCTCA, ATGCTCAG
reacción con ddGTP: ATG, ATGCTCAG
reacción con ddCTP: ATGC, ATGCTC, ATGCTCAG
reacción con ddTTP: AT, ATGCT, ATGCTCAG
todas las reacciones una Olwen reina):
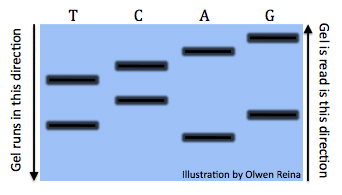
cada banda denota el código de diferentes longitudes. Por ejemplo, la banda está a la derecha debajo de la «A» simboliza la secuencia:»ATGCTCA»
todavía confundido?
imaginemos un juego de fiesta. El juego es un juego de adivinanzas., Así es como se juega:
estás pensando en un número y el grupo tiene que adivinarlo. La parte difícil es que el número es de 200 dígitos de longitud. Estás leyendo los dígitos del número en tu cabeza sin hacer un sonido. De vez en cuando una persona te interrumpe, y les dices el dígito que estabas pensando y dónde está en la secuencia de 200. Cada vez que te interrumpen, tienes que empezar de nuevo. Te vas después de unas horas y el grupo tiene que averiguar el número de 200 dígitos., Tienen que juntar la información que les diste, por ejemplo, el número 25 era 5, El número 40 era 0, y así sucesivamente. Usando la información de sus interrupciones, pueden repetir el número que te dieron.
mientras que esto suena como el juego más lamest en el mundo, funciona muy bien para la secuenciación!
desafortunadamente, es lento, costoso y (anteriormente) depende de materiales radiactivos. Esto empujó a los científicos a desarrollar nuevas y mejores formas de secuenciación del genoma.,
tecnologías de secuenciación de próxima generación
los mayores avances en la secuenciación del genoma han sido el aumento de la velocidad y la precisión, lo que resulta en una reducción en la mano de obra y el costo. La velocidad es gracias al análisis paralelo y la tecnología de alto rendimiento. Es la diferencia entre conseguir que un niño de 4 años lea Moby Dick y dar un párrafo cada uno a un montón de carreras de Drama y pedirles que lean a la vez.
Las máquinas de secuenciación han mejorado enormemente desde que Sanger desarrolló su método., En 1987, Applied Biosystems se convirtió en el PRIMERO en introducir una máquina de secuenciación automática, llamada AB370. La máquina se basó en un método llamado electroforesis capilar que utiliza el método de terminación de la cadena de Sanger sin necesidad de un gel.
ddNTPs de terminación de cadena marcados Fluorescentemente se añaden a la mezcla de reacción PCR. Mediante el método de secuenciación Sanger, se crean productos de PCR de varias longitudes y luego se separan de acuerdo con su tamaño. El tamaño se mide por la carga negativa total del producto PCR. Cuanto más negativa sea la carga, más largo será el fragmento., Tome el ejemplo anterior de incorporación de ddNTP e imagine que las letras en negrita son ddNTPs con un fluorocromo adjunto. A medida que los fragmentos son tirados hacia el electrodo positivo de un capilar (Ver imagen abajo), pasan un rayo láser que desencadena un destello de luz desde el fluorocromo unido al ddNTP que es característico del tipo base (por ejemplo, verde para A, amarillo para T, azul para G, rojo para C). De esta manera, el genoma se lee cuidadosamente.
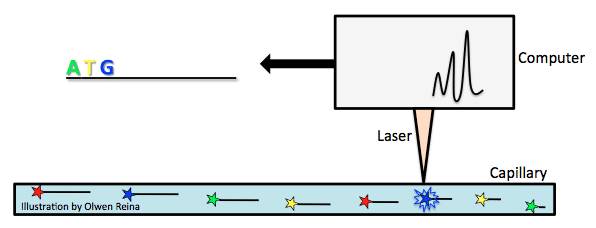
La mayor diferencia era la velocidad y el costo., El AB370 podía detectar 96 bases a la vez, 500k bases por día, y tenía una longitud de lectura de 600 bases mediante el uso de un análisis paralelo y una configuración de alto rendimiento. Esta forma de secuenciación se convirtió en la principal herramienta para la finalización del Proyecto Genoma Humano.
¿cómo se compara NGS con la secuenciación de primera generación?
NGS se caracteriza por mejorar la precisión y la velocidad, pero también por reducir la mano de obra y el costo. Nunca ha habido un tiempo en el que haya sido tan barato, conveniente o sencillo secuenciar un genoma., Podría decirse que la mayor mejora ha sido el desarrollo del análisis paralelo, que aumentó la velocidad de secuenciación.
¡lo único que nos ralentiza ahora es la interpretación de los resultados!

Tiene esto ha ayudado? Entonces por favor comparta con su red.
















Deja una respuesta